Juan Carlos de la Concepción Romero
Juan Francisco Rodríguez Alcázar
María Dolores Fernández Fernández
Mateo Seoane Blanco
Ignacio Campillo Marcos
José Antonio Valverde López

Describen cómo el interferón protege contra los hongos
Para luchar contra las infecciones, nuestro organismo cuenta con un tipo de moléculas conocidas como interferones que consiguen bloquear la multiplicación de los virus y activar a los glóbulos blancos que atacan tanto a estos como a las bacterias. En su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por Carlos Ardavín estudia el funcionamiento de unas células inmunitarias que producen interferón, las células dendríticas.
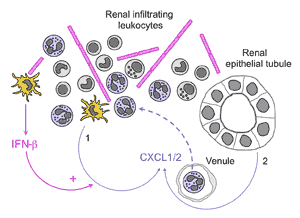 A parte del efecto contra virus y bacterias, hace años se descubrió que “las células dendríticas son clave en la inmunidad contra el hongo Candida albicans” explica Ardavín, quien acaba de publicar en la revista Immunity que para que puedan actuar contra dicho hongo es necesario que las células dendríticas produzcan interferón de tipo I (α y β). Sólo así, las células dendríticas reducen la mortalidad en infecciones causadas por Candida albicans en ratones.
A parte del efecto contra virus y bacterias, hace años se descubrió que “las células dendríticas son clave en la inmunidad contra el hongo Candida albicans” explica Ardavín, quien acaba de publicar en la revista Immunity que para que puedan actuar contra dicho hongo es necesario que las células dendríticas produzcan interferón de tipo I (α y β). Sólo así, las células dendríticas reducen la mortalidad en infecciones causadas por Candida albicans en ratones.
Profundizando en el mecanismo molecular, los científicos del CNB han descubierto el papel esencial de dos proteínas llamadas Dectin-1 y Dectin-2 para que las células dendríticas produzcan Interferón β cuando el organismo sufre una infección por dicho hongo. Sin ellas, no se produce el interferón y, entre otras cosas, los neutrófilos encargados de luchar contra la infección no se desplazan hasta el lugar donde se encuentra C. albicans.
- Del Fresno C, Soulat D, Roth S, Blazek K, Udalova I, Sancho D, Ruland J, Ardavín C. Interferon-β production via Dectin-1-Syk-IRF5 signaling in Dendritic Cells is crucial for immunity to C. albicans. Immunity 2013 Jun 27;38(6):1176-86.
Describen cómo la proteína AddAB recorre el ADN para su reparación
Produciéndose cerca de 10.000 alteraciones diarias en el genoma de cada célula, el trabajo que hacen proteínas reparadoras del ADN como las que estudia en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) el grupo dirigido por el biofísico Fernando Moreno-Herrero es tremendamente importante.
 Junto a bioquímicos de la Universidad de Bristol, han publicado en la revista PNAS el mecanismo de exploración o escaneo que utiliza la proteína AddAB en el proceso de reparación de un corte de la doble hélice de ADN. Como nos explica Moreno-Herrero, reparar los daños en esencial ya que su acumulación puede acabar provocando "la muerte celular o incluso degenerar en una célula cancerígena".
Junto a bioquímicos de la Universidad de Bristol, han publicado en la revista PNAS el mecanismo de exploración o escaneo que utiliza la proteína AddAB en el proceso de reparación de un corte de la doble hélice de ADN. Como nos explica Moreno-Herrero, reparar los daños en esencial ya que su acumulación puede acabar provocando "la muerte celular o incluso degenerar en una célula cancerígena".
Con anterioridad, el grupo del CNB había demostrado que para que la proteína AddAB recorriera el cromosoma separando las dos hebras necesita estar unida a unas secuencias del genoma llamadas Chi. Ahora además, utilizando la técnica de pinzas magnéticas, han sido capaces de estudiar molécula a molécula cómo recorre el ADN, a qué velocidad y hasta las pausas que hacen. Sus resultados muestran que las características de la translocación a lo largo del ADN dependen del contenido en citosina y guanina y de la presencia de las secuencias Chi.
Desde el CNB, la investigadora postdoctoral Carolina Carrasco nos explica que en su estudio han podido ver "que cuando la proteína encuentra una verdadera secuencia Chi, realiza una pausa en esta posición después de la cual reinicia nuevamente su trayectoria". Y es que cuando AddAB se encuentra con secuencias parecidas a Chi, también se detiene en un intento fallido de reconocer estas pseudo-Chi, siendo la duración de las pausas en estos casos menores.
Basándose en la capacidad de dicha proteína de reconocer específicamente las secuencias Chi, los investigadores han propuesto un modelo mecánico para explicar el origen de ambas pausas. La forma de la pausa y el aparente cambio de velocidad después de la misma actúan cómo un filtro selectivo cuando se encuentra ante una secuencia Chi genuina.
- Carrasco C, Gilhooly NS, Dillingham MS, Moreno-Herrero F. On the mechanism of recombination hotspot scanning during double-stranded DNA break resection. PNAS June 24, 2013.
¿Cuántas bacterias hay en tus manos?
 Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Las bacterias son un tipo de microbios tan pequeños que no los podemos ver a simple vista. Ni siquiera usando una lupa. Para poder verlas necesitamos un microscopio. Por eso, hasta que no se inventó el microscopio nadie sabía que existían.
Ahora sabemos que hay un montón de tipos distintos y conocemos muchas de las que viven cerca de nosotros: en el agua, en la tierra o dentro de nosotros mismos. Sí, incluso dentro de nuestras barrigas. Son minúsculas, pero todas juntas pesan casi tanto como un litro de leche.
Aunque no las podamos ver una a una, los científicos saben criarlas y tener tal cantidad de ellas que podemos ver los grupos que forman. A estos grupos, los científicos los llaman colonias.
Un científico llamado Julius Richard Petri inventó un sistema muy sencillo para criar las bacterias: una placa redonda en la que hay una gelatina que tiene los alimentos que las bacterias necesitan. ¿Cuántos grupos de bacterias (colonias) ves que han crecido en esta placa?.
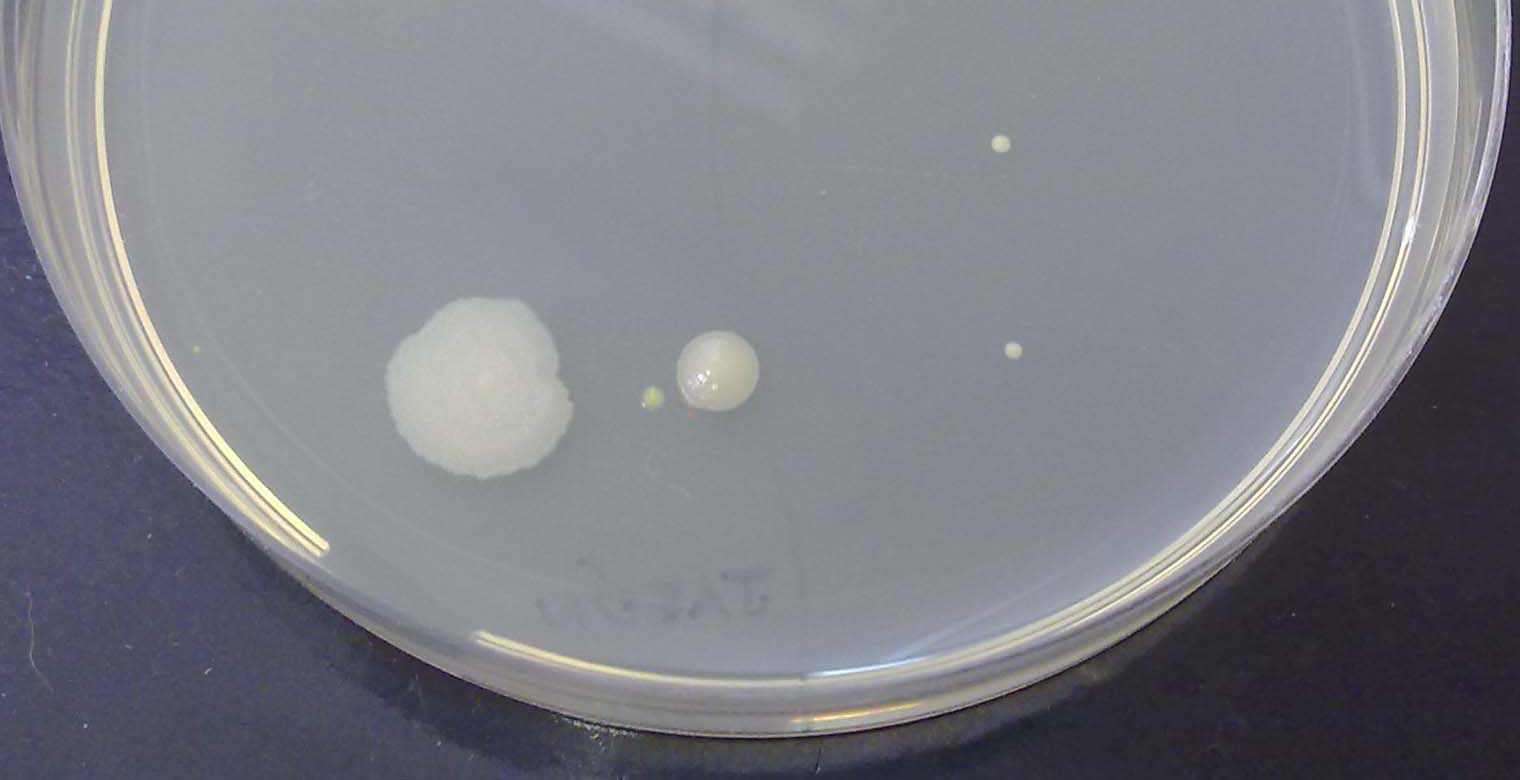
En nuestras manos podemos encontrar un montón de tipos distintos de bacterias. Para verlas, tan solo tenemos que tocar la gelatina de una de las placas de Petri y dejar que crezcan las colonias durante unos días. ¡Tócala con cuidado, no vaya a ser que se rompa!
Si lo haces con las manos limpias, ¿habrá más o menos bacterias? ¿Te apetece hacer el experimento? Toca una mitad de la placa con las manos sucias. Luego lávate las manos y toca la otra mitad. Según pasen los días, observa cómo crecen las colonias.
Como un buen científico, ve anotando durante los días siguientes cuantas colonias salen con las manos sucias y cuantas con ellas limpias.
El último día compara tus resultados con los del resto de la clase y pensar entre todos qué les pasa a las bacterias cuando te lavas las manos.
Una vez que hayan terminado todos los grupos, hay en poner en común los resultados obtenidos y sumar los números de todos y cada una de las parejas.
Esta práctica está basada en una visita que nos hicieron a principios de este año los alumnos del Colegio Rafael Alberti de Coslada. Si te interesa, esta sencilla práctica dirigida a alumnos educación primaria se puede descargar en pdf (2,37 MB).
Programa Internacional de doctorado La Caixa / CNB 2013
Por sexto año consecutivo, la Fundación "la Caixa" convoca su Programa Internacional de doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC.
 Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
En este sexto año consecutivo, la Fundación "la Caixa" convoca dos contratos predoctorales de personal investigador en formación para desarrollar un doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC, el mayor centro español y uno de los centros líderes en el mundo en el área de Biotecnología, Biología Molecular y Biomedicina.
Las bases de la convocatoria de este año, publicadas en el Boletín Oficial del Estado del 24 de junio de 2013, pueden descargarse como documento pdf (4,61 MB).
Los estudiantes deberán aportar la documentación acreditativa de estar admitido en un programa de doctorado expedido por el responsable de dicho programa.
Las solicitudes pueden enviarse hasta el 1 de julio de 2013.
- Listado provisional de admitidos y excluidos (subsanación de errores hasta el 13 de julio de 2013).
- Listado definitivo de admitidos y excluidos.
- Valoración provisional de méritos del concurso.
- Listado definitivo.
- Resolución definitiva.
España ya forma parte de la infraestructura europea de biología estructural
A través del Centro Nacional de Biotecnología del CSIC, que alberga la unidad de procesamiento de imágenes en microscopía electrónica líder en Europa, España se ha integrado en la mayor infraestructura europea de investigación en biología estructural (Instruct, en sus siglas en inglés).
 Gracias al acuerdo firmado entre el Consejo Superior de Investigaciones Científicas (CSIC) e Instruct, la comunidad científica española tendrá acceso a una red de infraestructuras científicas con capacidades en tecnologías clave para el estudio de la biología estructural de Alemania, Bélgica, Francia, Israel, Italia, Holanda, Portugal, Reino Unido y República Checa. Además, los españoles podrán participar activamente y de forma prioritaria en la preparación y puesta en marcha de nuevos proyectos europeos.
Gracias al acuerdo firmado entre el Consejo Superior de Investigaciones Científicas (CSIC) e Instruct, la comunidad científica española tendrá acceso a una red de infraestructuras científicas con capacidades en tecnologías clave para el estudio de la biología estructural de Alemania, Bélgica, Francia, Israel, Italia, Holanda, Portugal, Reino Unido y República Checa. Además, los españoles podrán participar activamente y de forma prioritaria en la preparación y puesta en marcha de nuevos proyectos europeos.
Los científicos españoles podrán participar activamente y acceder a múltiples infraestructuras Europeas para el desarrollo de sus proyectos de investigación. “El objetivo de esta iniciativa es unir los esfuerzos de varios países que comparten sus instalaciones para avanzar en biología y biomedicina. Los investigadores tendremos acceso a tecnologías de vanguardia, desde sincrotrones hasta grandes instalaciones de resonancia magnética nuclear”, ha señalado el investigador del CSIC José María Carazo.
El Centro Nacional de Biotecnología del CSIC coordina el proyecto en el ámbito del procesamiento de imagen en microscopía electrónica y una de sus misiones será involucrar a diversas instituciones y potenciar sinergias con otros centros de investigación e instalaciones singulares, como por ejemplo, el sincrotrón ALBA, el acelerador de partículas instalado en Cerdanyola del Vallès (Barcelona).
“Supone un gran reconocimiento internacional y nuestro compromiso será el desarrollar la nueva generación de infraestructuras en procesamiento de imagen en microscopía electrónica. Proporcionaremos apoyo a los biólogos experimentales y ayudaremos a maximizar la obtención de conocimiento biológico a partir de imágenes de microscopía. Es todo un honor y un reto el saber que la tecnología que diseñe nuestro grupo será la que se use a nivel europeo en el marco de Instruct”, ha precisado Carazo.
Instruct, la infraestructura europea de investigación en biología estructural, es una de las 48 que forman parte de la hoja de ruta del Foro Estratégico Europeo para las Infraestructuras de Investigación (ESFRI, en sus siglas en inglés). Estas grandes infraestructuras científicas internacionales constituyen la columna vertebral del Espacio Europeo de Investigación y son motor del desarrollo económico de los países involucrados en su construcción, favoreciendo la creación de economías más competitivas e impulsando la recuperación económica en momentos de crisis.
CNB workshop 'Division from A to Z'
7 June 2013
THE WORKSHOP: The divisome is an assembly of proteins involved in bacterial division. Some of its components, as FtsA and FtsZ, share structural and functional homologies with cytoskeletal proteins. Procedures to reconstruct divisomes in the test tube provide further insights on division and supply new discovery platforms to find new antibiotics.
The purpose of the workshop is to discuss the advances in the study of bacterial division.
KEYNOTE SPEAKER: From his postdoctoral at the University of Edinburgh to his present position at the University of Kansas Medical School, Professor Joe Lutkenhaus pioneering work has been crucial for the discovery and study of divisome components. He has received several academic recognitions, among them the prestigious Louisa Gross Horwitz Prize in 2012.
THE PROGRAMME: We have gathered Spanish microbiologists working on bacterial cell division and some investigators whose work, coming from fields different from microbiology, provide important advances to study bacterial division in the light of novel synthetic biology approaches.
VENUE: At the main CNB lecture room. C/ Darwin nº3. 28049 Madrid. Tel: +34 91 585 45 00
Attendance is free but room capacity is limited.
ORGANIZATION: Miguel Vicente (scientific) and Moira Torrent (technical)
WORKSHOP PROGRAMME (pdf)
10:00 - 10:30: José M. Valpuesta (CNB, Madrid) The folding pathway of actin and tubulin mediated by an assembly line of molecular chaperones
10:30 - 11:00: Luis M. Liz-Marzán (CIC biomaGUNE) Nanoplasmonic Biodetection
11:00 - 11:30: Miguel A. de Pedro (CBMSO, Madrid) Cell wall elongation and septation: Who leads whom?
11:30 - 12:00: Juan Ayala (CBMSO, Madrid) Endopeptidases and carboxypeptidases in bacterial cell cycle. How many and for what purpose?
12:00 -12:30 COFFEE BREAK
12:30 - 13:15: Joe Lutkenhaus (U. Kansas) Min system and the Z ring
13:15 - 13:45: Jesús Mingorance (Hospital La Paz, Madrid) FtsA, bacterial division and the quest for a role
14:00 - 15:30 LUNCH
15:30 - 16:00: Marisela Vélez (ICP, Madrid) Looking at individual FtsZ filaments on surfaces in solution: what we have learned so far
16:00 - 16:30: Germán Rivas (CIB, Madrid) Macromolecular interactions of FtsZ: from physical biochemistry to reconstructing minimal divisomes in cell-like environments
16:30 - 17:00: José M. Andreu (CIB, Madrid) Targeting bacterial cell division protein FtsZ with small molecules
17:00 - 17:30: Paulino Gómez-Puertas (CBMSO, Madrid) In silico design of new antimicrobial compounds directed against FtsZ
17:30 - 18:00: Francisco Monroy (U. Complutense, Madrid) Membrane mechanics of bacterial division: a connection to Z ring mechanistic
18:00 - 18:30: Miguel Vicente (CNB, Madrid) Zipping the Z
Identifican un gen clave en fertilidad y en el desarrollo de testículos
El grupo del Centro Nacional de Biotecnología del CSIC dirigido por Jesús M. Salvador acaba de publicar que la expresión del gen Gadd45g es crítica durante el desarrollo embrionario para la formación de testículos. Al generar ratones deficientes en la proteína Gadd45g se dieron cuenta de que sorprendentemente todas las crías eran fenotípicamente hembras.
 Aunque los ratones deficientes en Gadd45g tienen el cromosomas Y y por tanto genéticamente se considerarían machos, no desarrollan testículos por un problema en la señalización durante el desarrollo embrionario. La ausencia de Gadd45g origina una anomalía de la diferenciación sexual que ocasiona el desarrollo de ovarios independientemente de la presencia del cromosoma Y.
Aunque los ratones deficientes en Gadd45g tienen el cromosomas Y y por tanto genéticamente se considerarían machos, no desarrollan testículos por un problema en la señalización durante el desarrollo embrionario. La ausencia de Gadd45g origina una anomalía de la diferenciación sexual que ocasiona el desarrollo de ovarios independientemente de la presencia del cromosoma Y.
En humanos, las anomalías de la diferenciación sexual son un grupo amplio de patologías ocasionadas por diferentes alteraciones en alguna de las etapas del desarrollo fetal necesarias para el desarrollo normal del sexo genético y gonadal. Están consideradas dentro del grupo de patologías raras por su baja frecuencia inferior a 1/4.500 recién nacidos. La etiología de este tipo de patologías es genética y aunque se han descrito algunas mutaciones inactivadoras en un grupo muy reducido de genes, existe un gran número de casos sin diagnóstico definido. Por tanto la identificación de este gen de la familia Gadd45 puede ser muy importante en el esclarecimiento del diagnóstico etiológico de este tipo de patologías.
La familia de genes Gadd45, está compuesta por tres miembros, Gadd45a, Gadd45b y Gadd45g. En el laboratprio de Salvador han generado ratones deficientes en cada una de estas proteínas y observaron que Gadd45g es el único miembro de la familia que tiene una función esencial en la determinación del sexo y el desarrollo de testículos.
Además, en este estudio describen el mecanismo que ocasiona la anomalía en el desarrollo testicular. Gadd45g es un regulador positivo del gen SRY que se encuentra en el cromosoma Y. La ausencia de Gadd45g evita la expresión de SRY y SOX9, evitando el desarrollo de gónadas masculinas.
- Johnen H, González-Silva L, Carramolino L, Flores JM, Torres M, Salvador JM. Gadd45g is essential for primary sex determination, male fertility and testis development. PLoS ONE 8(3): e58751.
En la sombra las plantas tienen menos ramas
Las plantas, al contrario que los animales, son incapaces de ir de un sitio a otro en busca de alimento. Pero eso no les impide moverse e intentar de conseguir las condiciones de luz más favorables para ellas.
 Las plantas son capaces de percibir otras plantas en su proximidad y cuando esto sucede ponen en marcha un programa genético conocido como el síndrome de huida de la sombra. Un síndrome que, entre otras características, consiste en la supresión del crecimiento de las ramas laterales, lo que a su vez favorece el crecimiento del tallo principal. De ahí que las plantas que crecen muy juntas sean más altas que las que lo hacen disponiendo de más espacio.
Las plantas son capaces de percibir otras plantas en su proximidad y cuando esto sucede ponen en marcha un programa genético conocido como el síndrome de huida de la sombra. Un síndrome que, entre otras características, consiste en la supresión del crecimiento de las ramas laterales, lo que a su vez favorece el crecimiento del tallo principal. De ahí que las plantas que crecen muy juntas sean más altas que las que lo hacen disponiendo de más espacio.
En su laboratorio del Centro Nacional de Biotecnología del CSIC, Pilar Cubas ha descubierto que el gen BRANCHED1 se activa en las yemas axilares cuando hay plantas cercanas. Según acaba de publicar en la revista Plant Cell, al ponerse en funcionamiento este factor de transcripción TCP se encarga de reprimir la transcripción de un gran número de genes implicados tanto en el ciclo celular como en la síntesis de proteínas. Además, también promueve la acción de una hormona asociada a la dormición, el ácido abscísico. Como resultado, las yemas laterales entran en reposo y no crecen nuevas ramas. De este modo, la planta mejora su captación de luz y evita ser cubierta por las plantas que la rodean.
Conocer el funcionamiento de BRANCHED1 puede permitir en el futuro suprimir el crecimiento de ramas no deseadas en especies de interés agronómico en las que la poda supone un elevado coste de manejo y, explica Cubas, "mejorar la arquitectura de otras especies para favorecer su cosechado".
- González-Grandío E, Poza-Carrión C, Sorzano CO, Cubas P. BRANCHED1 promotes axillary bud dormancy in response to shade in Arabidopsis. Plant Cell. 2013 Mar;25(3):834-50.
1st CNB Course on Introduction to Research
The Centro Nacional de Biotecnología/CSIC (CNB) is organising its First Course on Introduction to Research for students in the 3rd and 4th years of any university degree in science. The course is designed for qualified and highly motivated students who would like to contact the scientists at the CNB. The goal of this course is to provide an overview of the various research techniques and expertise available at the CNB.
The four-week course will be held in July 2013 (1 – 26 July 2013).
In the first three weeks, the students will rotate through the various CNB departments, participating in all activities and seminars organised by each. The student will choose the department in which s/he will spend the last week of the course.
Selected Students
Laura Bernal Sánchez
Juan María Roldán Romero
Francisco José de Lamo Ruiz
Álvaro San Hipólito Luengo
Verónica Miguel Herranz
Raquel Patricia Puig Lozano
Jun Kyo Seo
Alba Rodríguez Meira
María Lorena Martínez Quiles
Ana Sánchez Cano
Antonio Lahera Alonso

Identifican un dominio que permite a las chaperonas unirse a distintas proteínas
Según se van sintetizando las proteínas en los ribosomas, éstas tienen que ir tomando la forma adecuada para poder ejercer su función. Aunque las características físico-químicas de los aminoácidos que las forman determinan en gran parte la forma que adquieren, hay muchas proteínas que necesitan ayuda extra de parte de un grupo de proteínas conocidas como chaperonas.
 Para comprender mejor cómo funcionan estas proteínas, en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por José María Valpuesta ha utilizado la microscopía electrónica. Gracias a esta técnica han podido determinar por primera vez la estructura de un complejo formado por la chaperona DnaJ y su sustrato, lo que les ha permitido observar cómo la chaperona cambia la estructura del sustrato y con ello su función.
Para comprender mejor cómo funcionan estas proteínas, en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por José María Valpuesta ha utilizado la microscopía electrónica. Gracias a esta técnica han podido determinar por primera vez la estructura de un complejo formado por la chaperona DnaJ y su sustrato, lo que les ha permitido observar cómo la chaperona cambia la estructura del sustrato y con ello su función.
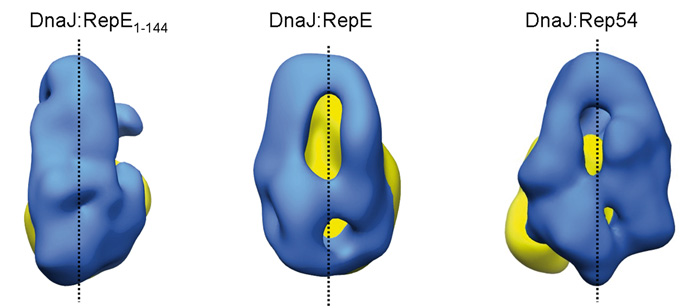
En colaboración con la Universidad del País Vasco, el investigador postdoctoral del CNB Jorge Cuéllar ha identificado además en dicha chaperona una zona de gran flexibilidad que le permite adaptarse a la forma de distintas proteínas. Como se puede apreciar en la imagen de abajo, la forma que adopta la chaperona DnaJ (en azul) cambia radicalmente en función del sustrato al que se une (RepE1-144, RepE o Rep54; en amarillo). De este modo, una misma chaperona es capaz de unirse a una variedad de proteínas diferentes, consiguiendo en todas ellas que adquieran la forma necesaria para funcionar.
- Cuéllar J, Perales-Calvo J, Muga A, Valpuesta JM, Moro F. Structural insights into the chaperone activity of the 40 kDa heat shock protein DnaJ. Binding and remodeling of a native substrate. J Biol Chem 2013 Apr 11. [doi: 10.1074/jbc.M112.430595].
