
Descubierta la estructura de la ribonucleoproteína del virus Bunyamwera
Un estudio internacional en el que ha participado la investigadora del CNB Cristina Risco ha publicado en la revista PNAS la estructura de la ribonucleoproteína del virus Bunyamwera, modelo de estudio de los integrantes de la familia Bunyaviridae, a la que también pertenecen los virus que provocan la fiebre hemorrágica de Congo y Crimea y la fiebre del valle del Rift.
"Uno de los principales resultados de esta investigación es que la flexibilidad de la nucleoproteína que envuelve el ARN de Bunyamwera facilita el empaquetamiento de su material genético en las partículas virales y su replicación una vez fuera del virus. La estructura atómica muestra que el plegamiento de la nucleoproteína es, además, diferente al observado en las demás nucleoproteínas de virus conocidas”, explica Risco.
Este estudio muestra que, a pesar de su flexibilidad, el complejo que recubre el material genético lo protege completamente del ataque de las nucleasas, las enzimas que rompen las moléculas de ARN.
Los resultados de este estudio ayudan a entender mejor el mecanismo molecular mediante el que se ensamblan las ribonucleoproteínas virales. "Este conocimiento permitirá avanzar en el desarrollo de compuestos para atacar a estos complejos macromoleculares que son esenciales para la supervivencia de los virus ARN", añade desde el mismo laboratorio del Centro Nacional de Biotecnología del CSIC la investigadora Isabel Fernández de Castro.
Los hallazgos logrados por este estudio, liderado por investigadores chinos, ha sido posible mediante el uso de cristalografía de rayos X y microscopía electrónica. Como se aprecia en la imagen, el complejo formado por el monómero de la proteína viral NP y el ARN, la flexibilidad de este complejo respecto a los adyacentes es el elemento básico que dirige la oligomerización y ensamblaje de las ribonucleoproteínas lineales y flexibles del virus Bunyamwera.
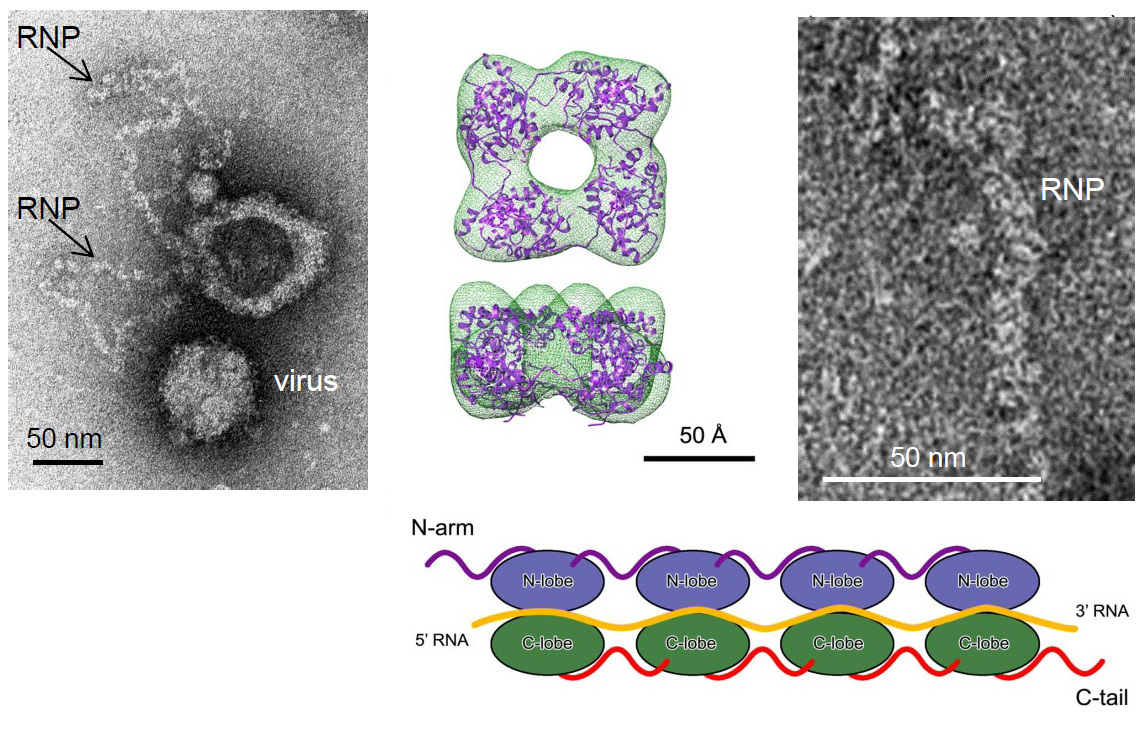
- Li B, Wang Q, Pan X, Fernández de Castro I, Sun Y, Guo Y, Tao X, Risco C, Su SF, Lou Z. Bunyamwera virus possesses a distinct nucleocapsid protein to facilitate genome encapsidation. PNAS April 8, 2013 doi: 10.1073/pnas.1222552110.
Premio de Investigación CEI UAM+CSIC en Biología Molecular Física y Sintética
El Campus de Excelencia Internacional UAM+CSIC (CEI UAM+CSIC) tiene como una de sus líneas de actuación esenciales la captación de talento tanto a nivel nacional como internacional. Y con la intención de atraer científicos y profesores de primer nivel, acaba de convocar el Premio de Investigación CEI UAM+CSIC en el área de la Biología Molecular Física y Sintética.
 El objetivo de este premio, compatible con cualquier otro tipo de premio y/o ayuda, es el de favorecer que profesionales de otros centros de investigación puedan trasladarse al CNB para desarrollar una línea de investigación que se centre en el estudio del plegamiento y función de proteínas. Para ello, deberán integrar métodos experimentales de alta resolución temporal, estructural y de moléculas únicas, modelización teórica y simulaciones computacionales.
El objetivo de este premio, compatible con cualquier otro tipo de premio y/o ayuda, es el de favorecer que profesionales de otros centros de investigación puedan trasladarse al CNB para desarrollar una línea de investigación que se centre en el estudio del plegamiento y función de proteínas. Para ello, deberán integrar métodos experimentales de alta resolución temporal, estructural y de moléculas únicas, modelización teórica y simulaciones computacionales.
El premio tiene una dotación económica de 30.000 € anuales de libre disposición durante tres años, prorrogables a otros dos. Para solicitarlo, el investigador debe tener una vinculación permanente con el CSIC y no podrá estar adscrito a ningún centro del CEI UAM+CSIC.
Las solicitudes se presentarán en el registro general de la UAM desde el 8 de abril al 22 de abril de 2013.
La Reina Doña Sofía visitó el CNB
Con motivo del vigésimo aniversario del Centro Nacional de Biotecnología del CSIC (CNB), la semana pasada, la Reina Doña Sofía visitó sus laboratorios en compañía de la directora del CNB, Carmen Castresana, y de su antecesor en el cargo, José María Valpuesta.
 Doña Sofía, acompañada, entre otras autoridades, por la ministra de Fomento, Ana Pastor, el presidente del CSIC, Emilio Lora-Tamayo, y el rector de la Universidad Autónoma de Madrid, José Mª Sanz Martínez, mantuvo una reunión de trabajo con el director saliente, José María Valpuesta; su sucesora, Carmen Castresana; tres investigadores que también ocuparon este puesto (Mariano Esteban, José Ramón Naranjo y José López Carrascosa); y el científico del CNB Carlos Martínez-Alonso, quien fuera en su momento presidente del CSIC y anterior Secretario de Estado de Investigación.
Doña Sofía, acompañada, entre otras autoridades, por la ministra de Fomento, Ana Pastor, el presidente del CSIC, Emilio Lora-Tamayo, y el rector de la Universidad Autónoma de Madrid, José Mª Sanz Martínez, mantuvo una reunión de trabajo con el director saliente, José María Valpuesta; su sucesora, Carmen Castresana; tres investigadores que también ocuparon este puesto (Mariano Esteban, José Ramón Naranjo y José López Carrascosa); y el científico del CNB Carlos Martínez-Alonso, quien fuera en su momento presidente del CSIC y anterior Secretario de Estado de Investigación.
Durante la reunión, Naranjo expuso los pormenores de su proyecto de investigación sobre el alzheimer que se financia, en parte, gracias a la Fundación Reina Sofía. Mariano Esteban tuvo ocasión asimismo de exponer a la Reina los avances obtenidos en los ensayos clínicos de su vacuna contra el sida y sus experimentos con vacunas contra la malaria, la leishmaniasis y la gripe. López Carrascosa, por su parte, explicó la marcha de sus trabajos sobre la estructura de los virus. Finalmente Martínez-Alonso habló de sus estudios sobre el papel de las células madre en el desarrollo y la metástasis del cáncer.
 A continuación realizaron un recorrido por las instalaciones del CNB, entre las que destacan el servicio de microscopía confocal, el microscopio electrónico, el citómetro de flujo, el laboratorio de pinzas ópticas y las instalaciones de cultivo de plantas del CNB.
A continuación realizaron un recorrido por las instalaciones del CNB, entre las que destacan el servicio de microscopía confocal, el microscopio electrónico, el citómetro de flujo, el laboratorio de pinzas ópticas y las instalaciones de cultivo de plantas del CNB.
La visita comenzó en el laboratorio de microscopía confocal. En el, Sylvia Gutiérrez Erlandsson mostró las imágenes digitales que se obtienen actualmente de las células gracias al empleo de láseres. Con esta tecnología y el uso de marcadores fluorescentes en las muestras, se pueden localizar los distintos componentes de las células.
Durante la visita al microscopio electrónico, Jaime Martín-Benito le pudo explicar cómo funcionan este tipo de microscopios y le estuvo enseñando algunos ejemplos como su último trabajo publicado en la revista Science en el que describen la estructura de las proteínas responsables de la replicación y expresión del material genético del virus de la gripe A.
 A continuación, María del Carmen Moreno-Ortiz explicó a Doña Sofía en qué consiste la técnica de la citometría de flujo. Entre los ejemplos interesantes de cómo se pueden analizar las distintas poblaciones celulares de una muestra, Moreno tuvo la oportunidad de mostrar cómo se pueden separar las células tumorales de las células normales.
A continuación, María del Carmen Moreno-Ortiz explicó a Doña Sofía en qué consiste la técnica de la citometría de flujo. Entre los ejemplos interesantes de cómo se pueden analizar las distintas poblaciones celulares de una muestra, Moreno tuvo la oportunidad de mostrar cómo se pueden separar las células tumorales de las células normales.
Su Majestad tuvo la oportunidad de conocer de mano de Elías Herrero y Borja Ibarra la llamada técnica de pinzas ópticas. Esta revolucionaria técnica permite a los investigadores atrapar cualquier molécula individualmente (desde una proteína al ADN) y medir los cambios que sufre mientras ejerce su función.
Finalmente, en el invernadero del CNB, Carlos Alonso y Salomé Prat tuvieron la oportunidad de mostrar brevemente las plantas que se cultivan y explicar las últimas investigaciones llevadas a cabo por los científicos del Departamento de Genética Molecular de Plantas.

Mariano Esteban se incorpora al Comité Científico Asesor del CSIC
El CSIC ha renovado el panel de integrantes de su Comité Científico Asesor. La nueva lista de 23 miembros se ha cerrado con siete nuevas incorporaciones. Entre otras la de investigador del Centro Nacional de Biotecnología Mariano Esteban.
 El órgano, cuya presidencia ostenta el presidente del CSIC, Emilio Lora-Tamayo, se compone de científicos y tecnólogos de las distintas áreas de conocimiento en las que está distribuida la actividad científica del CSIC. Según el Estatuto del organismo, tiene la función de informar y asesorar en aspectos científico-tecnológicos a la Presidencia del CSIC y al Consejo Rector.
El órgano, cuya presidencia ostenta el presidente del CSIC, Emilio Lora-Tamayo, se compone de científicos y tecnólogos de las distintas áreas de conocimiento en las que está distribuida la actividad científica del CSIC. Según el Estatuto del organismo, tiene la función de informar y asesorar en aspectos científico-tecnológicos a la Presidencia del CSIC y al Consejo Rector.
Los siete vocales que se incorporan al comité son: Juan Albadalejo Montoro (profesor de Investigación del CSIC adscrito al Centro de Edafología y Biología Aplicada del Segura), Mariano Esteban Rodríguez (profesor de Investigación del CSIC adscrito al Centro Nacional de Biotecnología), Ángel Messeguer Peypoch (profesor de Investigación del CSIC adscrito al Instituto de Química Avanzada de Cataluña) y Juan Moreno Klemming (profesor de Investigación del CSIC adscrito al Museo Nacional de Ciencias Naturales)
Esteban es pionero en el campo de las vacunas, donde destaca en la lucha contra el sida empleando procedimientos de inmunización combinada de vectores. Sus trabajos, que están siendo financiados por distintos organismos nacionales e internacionales como la Fundación Bill y Melinda Gates, tienen aplicación en enfermedades como la hepatitis C, la gripe o el cáncer de próstata.
La Unión Europea concede una Advanced Grant a Víctor de Lorenzo
Una de las 3 Advanced Grants en el campo de las Ciencias de la Vida que la Unión Europea ha concedido este año a científicos españoles será para el investigador del Centro Nacional de Biotecnología del CSIC (CNB) Víctor de Lorenzo. Con la participación de otro científico de la casa, Luis Ángel Fernández, desarrollarán un gran proyecto de Biología Sintética.
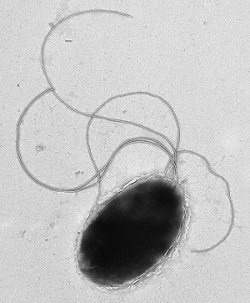 Víctor de Lorenzo es un ingeniero genético especializado en biorremediación ambiental y creó hace unos años una bacteria que emite luz en presencia de residuos de explosivos. Ahora recibirá de la Comisión Europea durante los próximos 5 años la financiación necesaria para la creación de un sistema inmune artificial.
Víctor de Lorenzo es un ingeniero genético especializado en biorremediación ambiental y creó hace unos años una bacteria que emite luz en presencia de residuos de explosivos. Ahora recibirá de la Comisión Europea durante los próximos 5 años la financiación necesaria para la creación de un sistema inmune artificial.
El objetivo del proyecto, explica de Lorenzo, es "la creación de una plataforma artificial basada exclusivamente en partes, dispositivos y módulos de origen bacteriano". La construcción de este sistema en bacterias tiene como intención simplificar su generación y extender las aplicaciones de los anticuerpos más allá del ámbito biomédico.
El proyecto explotará los conceptos de diseño, las jerarquías de construcción y las nociones de estandarización que se derivan de las corrientes más actuales de la Biología Sintética. El resultado será el ensamblaje y validación de lo que creen que será el sistema biológico no-natural más complejo intentado hasta el momento.
Mejoran en el CNB los anticuerpos humanizados producidos por ratones
El laboratorio del investigador del Centro Nacional de Biotecnología del CSIC (CNB) Lluís Montoliu ha generado para la empresa británica Crescendo Biologics Limited una variedad de ratones con anticuerpos humanos mejores que los que se producían hasta ahora.
 Los anticuerpos humanos se llevan usando hace ya bastantes años para el tratamiento de enfermedades como la artritis reumatoide o la enfermedad de Crohn. Ahora, con la tecnología diseñada por Crescendo se ha dado un paso más y los ratones generados tan sólo producen una de las partes de los anticuerpos, la llamada cadena pesada variable.
Los anticuerpos humanos se llevan usando hace ya bastantes años para el tratamiento de enfermedades como la artritis reumatoide o la enfermedad de Crohn. Ahora, con la tecnología diseñada por Crescendo se ha dado un paso más y los ratones generados tan sólo producen una de las partes de los anticuerpos, la llamada cadena pesada variable.
Estos anticuerpos, mucho más pequeños que los naturales, tienen la ventaja de poder usarse vía tópica y, según comenta Mike Romanos, director ejecutivo de Crescendo Biologics, “llegan con más facilidad a los órganos y tejidos en los que deben actuar y son más baratos de producir a gran escala”.
Desde el laboratorio del CNB en el que se han generado esta variedad de ratones, Lluís Montliu nos explica que el éxito de estos nuevos ratones transgénicos que producen los segmentos pesados de anticuerpos humanos se pone de manifiesto al usar ratones inmunodeficientes, que carecen de anticuerpos propios, propiedad de la empresa, permitiendo la obtención de los anticuerpos humanos sin contaminación alguna con anticuerpos de ratón. Un nuevo tipo de anticuerpos terapéuticos que Crescendo quiere empezar a utilizar en colaboración con empresas farmacéuticas.
Mariano Esteban es elegido Presidente de la Real Academia Nacional de Farmacia
El investigador del Centro Nacional de Biotecnología del CSIC (CNB) Mariano Esteban sustituye a María Teresa Miras Portugal en la presidencia de la Real Academia Nacional de Farmacia. Y lo hace con la “ilusión de impulsar desde la Academia todo lo relacionado con el medicamento y la salud”, especialmente a través de conferencias, mesas redondas y simposios dirigidos a entender los mecanismos moleculares de la acción de los fármacos sobre el organismo.
 Durante la Sesión Inaugural del curso del pasado jueves 17 de enero, Esteban tomó posesión como nuevo presidente de la institución. Desde su nuevo cargo anima a que en una época en la que la biología avanza a pasos agigantados, se apliquen esos conocimientos “para un mayor bienestar social”.
Durante la Sesión Inaugural del curso del pasado jueves 17 de enero, Esteban tomó posesión como nuevo presidente de la institución. Desde su nuevo cargo anima a que en una época en la que la biología avanza a pasos agigantados, se apliquen esos conocimientos “para un mayor bienestar social”.
Pionero en el campo de las vacunas, caben destacar sus investigaciones en la lucha contra el sida empleando procedimientos de inmunización combinada de vectores. Sus trabajos, que están siendo financiados por distintos organismos nacionales e internacionales como la Fundación Bill y Melinda Gates, tienen aplicación en enfermedades como la hepatitis C, la gripe o el cáncer de próstata.
Para el que fuera director del CNB de 1992 a 2003, la Academia debe “fomentar el estudio de las llamadas enfermedades olvidadas”. Algo a lo que el mismo ha dedicado gran parte de su vida científica. No en vano, el propio Esteban cuenta con más de 270 artículos en revistas internacionales relacionados no solo con la producción de vacunas contra el VIH sino también contra enfermedades como la leishmaniasis o la malaria.
Para este licenciado en Farmacia (1967) y en Ciencias Biológicas (1972), la Academia “debe ser un foro donde brillen las ideas” y recalca la importancia de seguir colaborando con el sector farmacéutico ya que ambos se necesitan mutuamente.
XX Jornadas Científicas de Navidad del CNB
Descrita la estructura de la ribonucleoproteína del virus de la gripe A
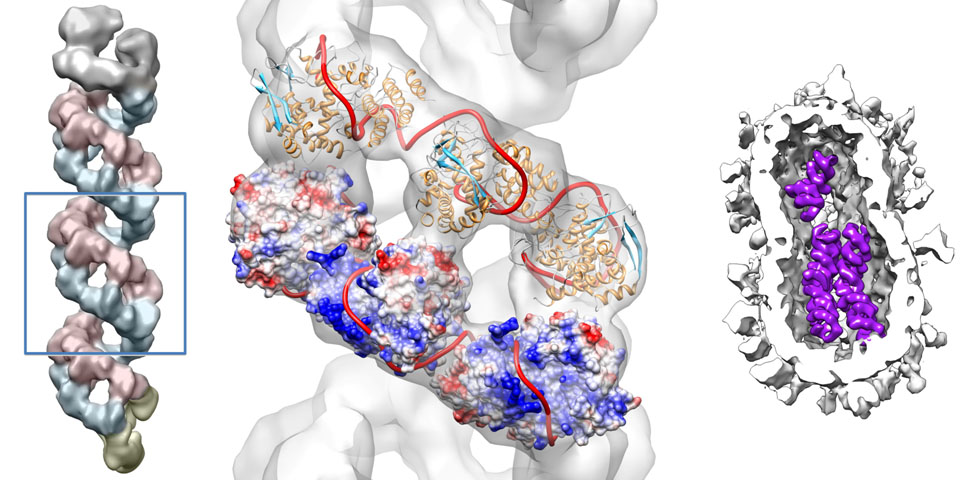
Bajo la dirección de Jaime Martín-Benito y Juan Ortín, se acaba de publicar en la revista Science la estructura de las proteínas del virus de la gripe responsables de la replicación y expresión de su material genético.
 Los virus de la gripe A son endémicos en aves y también infectan al hombre y otros mamíferos, causando epidemias anuales y ocasionalmente pandemias que constituyen un grave problema sanitario. Estas epidemias ocasionan un exceso de mortalidad, generalmente en personas de avanzada edad o con su inmunidad debilitada. Además, la amenaza de pandemias de muy alta virulencia sigue siendo un peligro real, como muestran los datos de mortalidad de algunas cepas como la conocida “gripe aviar”. Estos virus de la gripe, del subtipo H5N1, no se transmiten de forma directa entre humanos, pero presentan unas tasas de mortalidad de casi el 60% (359 muertes de 608 casos registrados por la Organización Mundial de la Salud desde 2003 hasta agosto de 2012). La importancia de este patógeno también radica en el enorme impacto económico que tiene, tanto en los gastos directos sanitario-farmacéuticos como en la pérdida de decenas de millones de horas laborales en el mundo, ya que puede llegar a suponer hasta el 12% de las bajas laborables en invierno.
Los virus de la gripe A son endémicos en aves y también infectan al hombre y otros mamíferos, causando epidemias anuales y ocasionalmente pandemias que constituyen un grave problema sanitario. Estas epidemias ocasionan un exceso de mortalidad, generalmente en personas de avanzada edad o con su inmunidad debilitada. Además, la amenaza de pandemias de muy alta virulencia sigue siendo un peligro real, como muestran los datos de mortalidad de algunas cepas como la conocida “gripe aviar”. Estos virus de la gripe, del subtipo H5N1, no se transmiten de forma directa entre humanos, pero presentan unas tasas de mortalidad de casi el 60% (359 muertes de 608 casos registrados por la Organización Mundial de la Salud desde 2003 hasta agosto de 2012). La importancia de este patógeno también radica en el enorme impacto económico que tiene, tanto en los gastos directos sanitario-farmacéuticos como en la pérdida de decenas de millones de horas laborales en el mundo, ya que puede llegar a suponer hasta el 12% de las bajas laborables en invierno.
Un paso importante en el ciclo de vida del virus es la replicación y expresión de su material genético. El mensaje genético del virus está codificado en 8 segmentos de ácido ribonucleico (ARN). El ARN está unido a 4 proteínas diferentes: la polimerasa (compuesta por tres subunidades), que mantiene unidos los extremos del segmento, y múltiples copias de una misma proteína llamada nucleoproteína, que se unen al ARN como si fueran las cuentas de un collar. Esta compleja estructura es conocida como ribonucleoproteína (RNP) y funciona realmente como una máquina molecular, capaz de transcribir el mensaje genético del virus y de autoreplicarse dentro de la célula infectada para así generar la progenie de nuevos virus que infectarán otras células. El conjunto de las 8 RNPs que contienen toda la información genética del virus está empaquetado dentro de cada partícula viral de forma ordenada.
A pesar de su importancia, la información detallada de cómo se organizaban las proteínas y el ARN para generar estas RNPs era muy escasa. Por este motivo, y durante los últimos años, los laboratorios del CNB dirigidos por Juan Ortín, también investigador del CIBER de Enfermedades Respiratorias (CIBERES), y Jaime Martín-Benito han desarrollado un proyecto para la determinación de dicha estructura. La metodología de trabajo consistió en la extracción y purificación de las 8 RNPs presentes en las partículas virales, para posteriormente estudiarlas mediante técnicas de microscopía electrónica y procesamiento computacional de imágenes. Ese procedimiento permitió determinar la estructura de las RNPs aisladas y desvelar la organización de las proteínas que forman esta compleja máquina molecular. Finalmente, y usando técnicas de tomografía electrónica sobre virus intactos, se verificó que la estructura obtenida de las RNPs aisladas era la misma que la que se encontraba dentro del virus infectivo.
La estructura final obtenida muestra una organización de doble hélice con dos cadenas de ARN-nucleoproteína y la polimerasa situada en uno de los extremos del helicoide. Adicionalmente se ha verificado que dentro del virus intacto las diferentes RNPs estan dispuestas formando haces muy estrechamente empaquetados. El análisis de estos resultados, junto con los datos previos existentes, permitirá desvelar algunos de los pasos cruciales en el ciclo de vida del virus tales como los mecanismos por los que las RNPs se replican y expresan la información genética del virus, el modo en que las 8 RNPs se asocian entre sí en la partícula del virus o los mecanismos por los que los genes de virus aviares pueden transferirse a los virus humanos.
- Arranz R, Coloma R, Chichón FJ, Conesa JJ, Carrascosa JL, Valpuesta JM, Ortín J, Martín-Benito J. The structure of native influenza virion ribonucleoproteins. Science doi: 10.1126/science.1228172
¿Podemos atrapar células usando luz?
Con motivo de la Semana de la Ciencia 2012, desde el laboratorio de Nanomanipulación Óptica del IMDEA Nanociencia y del Centro Nacional de Biotecnología del CSIC, Elías Herrero Galán y Ricardo Arias González van a retransmitir vía Twitter (@CNB_CSIC) un experimeto: la captura y manipulación de células en un dispositivo de pinzas ópticas.
 La técnica de pinzas ópticas permite la captura y manipulación de partículas desde unos pocos nanómetros a centenas de micras. Mediante la aplicación de campos electromagnéticos, estas partículas no solo se pueden mover, sino que es posible medir pequeños desplazamientos en el rango de los nanómetros y fuerzas aplicadas sobre ellas en la escala de los piconewtons.
La técnica de pinzas ópticas permite la captura y manipulación de partículas desde unos pocos nanómetros a centenas de micras. Mediante la aplicación de campos electromagnéticos, estas partículas no solo se pueden mover, sino que es posible medir pequeños desplazamientos en el rango de los nanómetros y fuerzas aplicadas sobre ellas en la escala de los piconewtons.
En el Laboratorio de Nanomanipulación Óptica del IMDEA Nanociencia y del Centro Nacional de Biotecnología disponemos de un dispositivo capaz de manipular materia de esas características mediante la cofocalización de dos haces láser de igual intensidad que generan una trampa óptica simétrica.
Con el fin de comprobar si podemos emplear este aparato para realizar experimentos con células vivas, intentaremos fluir células, tanto eucariotas como procariotas, para estudiar cómo se comportan en presencia de estos campos electromagnéticos y determinar si es posible atraparlas y manipularlas sin comprometer su integridad.
El próximo miércoles día 14 de noviembre, iremos retransmitiendo el experimento en la cuenta de twitter del CNB.
Acabamos de llegar al laboratorio. Lo primero que tenemos que hacer es encender las pinzas ópticas twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Mientras se estabiliza la temperatura de los láseres montamos la cámara de fluidos, en la que introducimos las células twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012La cámara de fluidos son dos láminas adherentes silueteadas con la forma de los canales y selladas entre dos cubres twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012La cámara de fluidos también dispone de dos tubos conectores entre canales y una micropipeta que llega al canal central twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Colocamos la cámara entre los objetivos, fluimos tampón y alineamos dos láseres para maximizar la potencia de la trampa twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Ya estamos listos para empezar a fluir células y tratar de atraparlas bit.ly/XEqYNT
— CNB (@CNB_CSIC) noviembre 14, 2012Empezaremos por células eucariotas, que al ser más grandes resultarán más sencillas de encontrar y de atrapar bit.ly/XEqYNT
— CNB (@CNB_CSIC) noviembre 14, 2012Disponemos de células COS-7 en cultivo, que resuspendemos y diluimos antes de fluir en la cámara de las pinzas twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012A través de una jeringa conectada a tubos de polietileno, fluimos las células en su medio de cultivo por el canal superior…
— CNB (@CNB_CSIC) noviembre 14, 2012…y esperamos que lleguen al canal central a través de uno de los tubos conectores.
— CNB (@CNB_CSIC) noviembre 14, 2012Parece que no llega ninguna célula a la zona de captura. Quizás son demasiado grandes para el diámetro del tubo...
— CNB (@CNB_CSIC) noviembre 14, 2012Vamos a probar a fluirlas directamente por el canal central, pero diluiremos un poco más para evitar tener demasiadas twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Parece que ahora sí llegan. ¡Aquí tenemos una! Tiene un diámetro de unas diez micras twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012¡La acercamos a la trampa óptica y se atrapa! Parece que lo hace a través del núcleo, que es la zona de mayor densidad twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012No se observan efectos negativos visibles en la célula después de un tiempo en la trampa, aunque es imposible saber qué pasa en su interior
— CNB (@CNB_CSIC) noviembre 14, 2012La soltamos y la volvemos a atrapar sin aparente daño
— CNB (@CNB_CSIC) noviembre 14, 2012Ahora vamos a tratar de arrastrarla hasta la pipeta twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Hemos podido traerla hasta la pipeta sin dificultad. Veamos si somos capaces de succionarla...
— CNB (@CNB_CSIC) noviembre 14, 2012Perfecto. La célula ha quedado inmovilizada en la pipeta sin problemas y sin sufrir deformación de su membrana twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Con la célula inmovilizada en la pipeta ahora podríamos aproximarle sustratos con la trampa y estudiar su comportamiento mecánico
— CNB (@CNB_CSIC) noviembre 14, 2012Veamos ahora qué sucede con células procariotas, más pequeñas. Pero primero hay que fluir agua en la cámara para limpiarla
— CNB (@CNB_CSIC) noviembre 14, 2012La celda está lista para un nuevo experimento, pero antes vamos a hacer una pausa para comer. Nos vemos en media hora twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012De vuelta a las pinzas twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Disponemos de un cultivo en medio líquido de Escherichia coli, que diluiremos como hemos hecho antes para las COS-7 twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Fluimos ahora las bacterias directamente por el canal central
— CNB (@CNB_CSIC) noviembre 14, 2012Aquí llega una. Le aproximamos la trampa… ¡y la atrapamos sin problemas! bit.ly/UGpmgt
— CNB (@CNB_CSIC) noviembre 14, 2012En este caso, la bacteria se orienta en la dirección de propagación del láser, como si cada haz atrapara un extremo twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012¡Vaya! La bacteria es demasiado pequeña para succionarla con la pipeta, pero ¿podremos orientarla desplazando un haz con respecto del otro?
— CNB (@CNB_CSIC) noviembre 14, 2012Lentamente, desplazamos un haz láser lateralmente… ¡y vemos cómo la bacteria se orienta en esa dirección! twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Podemos orientar la bacteria a nuestro antojo tanto en vertical como en horizontal desplazando el láser twitter.com/CNB_CSIC/statu…
— CNB (@CNB_CSIC) noviembre 14, 2012Tampoco se observa ningún daño aparente en la bacteria tras un tiempo en la trampa
— CNB (@CNB_CSIC) noviembre 14, 2012Interesantes resultados... Ahora toca repetirlos durante la tarde con diferentes células para ver si se reproduce el mismo comportamiento
— CNB (@CNB_CSIC) noviembre 14, 2012Es todo. Esperamos que os haya resultado interesante. Nosotros, a seguir trabajando
— CNB (@CNB_CSIC) noviembre 14, 2012