
Un científico del CNB, entre los más citados del mundo
 En su listado de los científicos más influyentes del mundo Thomson Reuters ha incluido a Roberto Solano. Este biólogo del Centro Nacional de Biotecnología del CSIC es uno de los 47 investigadores españoles que se encuentran entre el 1% más destacado de los científicos más citados del mundo en sus respectivas áreas de trabajo. A Solano podemos encontrarle junto a los 176 científicos que Thomson Reuters incluye en el área de Ciencias Animales y de Plantas.
En su listado de los científicos más influyentes del mundo Thomson Reuters ha incluido a Roberto Solano. Este biólogo del Centro Nacional de Biotecnología del CSIC es uno de los 47 investigadores españoles que se encuentran entre el 1% más destacado de los científicos más citados del mundo en sus respectivas áreas de trabajo. A Solano podemos encontrarle junto a los 176 científicos que Thomson Reuters incluye en el área de Ciencias Animales y de Plantas.
Solano estudia en su laboratorio los mecanismos moleculares que permiten a las plantas adaptarse al medio ambiente, centrándose en el funcionamiento del jasmonato, una hormona imprescindible para responder al estrés. Sus trabajos han llevado al descubrimiento de la forma activa de esta hormona y al de una familia de proteínas imprescindibles para que el jasmonato ejerza su acción. Estos dos descubrimientos, publicados en las revistas Nature y Nature Chemical Biology, han sido ya citados por otros investigadores más de 750 veces.
El número de citas de un trabajo es un indicador tanto del interés como de la influencia de dicho estudio para otros investigadores. Además, el prestigio de formar parte de la lista de Highly Cited Researchers también recae sobre las instituciones en las que se desarrollan los proyectos científicos. Estos datos los emplea el conocido como Ranking de Shanghái para valorar a las universidades y centros de investigación.
Además de Solano, otros 12 científicos del CSIC se encuentran incluidos en este prestigioso listado.
Descubren por qué la sombra reduce las defensas de las plantas
Investigadores del Centro Nacional de Biotecnología del CSIC (CNB) han descubierto el mecanismo por el que la sombra reduce las defensas de las plantas. Este descubrimiento puede ayudar a diseñar estrategias biotecnológicas que mejoren la producción agrícola en condiciones de alta densidad de cultivo.
 La alta densidad de cultivos en la agricultura moderna es responsable del aumento de productividad de las explotaciones agrarias. Sin embargo, tiene un efecto negativo sobre la capacidad de las plantas de defenderse contra insectos y otros patógenos. Según explica el investigador del CNB Roberto Solano, “la sombra que genera la alta densidad de cultivo actúa como una señal que induce el crecimiento vegetal para alcanzar la luz, pero esa redistribución de recursos para el crecimiento tiene como contrapartida una disminución de la capacidad de activación de defensas, entre ellas las que dependen de la fitohormona jasmonato”.
La alta densidad de cultivos en la agricultura moderna es responsable del aumento de productividad de las explotaciones agrarias. Sin embargo, tiene un efecto negativo sobre la capacidad de las plantas de defenderse contra insectos y otros patógenos. Según explica el investigador del CNB Roberto Solano, “la sombra que genera la alta densidad de cultivo actúa como una señal que induce el crecimiento vegetal para alcanzar la luz, pero esa redistribución de recursos para el crecimiento tiene como contrapartida una disminución de la capacidad de activación de defensas, entre ellas las que dependen de la fitohormona jasmonato”.
El grupo dirigido por Solano acaba de publicar un trabajo en la revista Plant Cell en el que identifican por primera vez los factores de transcripción necesarios para activar las defensas de la planta frente a insectos y patógenos necrótrofos. En el laboratorio del CNB, José Manuel Chico ha descubierto que los activadores transcripcionales MYC2, MYC3 y MYC4 están regulados negativamente por los represores JAZ. En condiciones de luz, los fotorreceptores de la planta (principalmente PhyB) mantienen niveles altos de MYC2, MYC3 y MYC4. Sin embargo, la sombra inactiva a PhyB y eso reduce la estabilidad de estos activadores transcripcionales, reduciendo los niveles de estas proteínas. Al mismo tiempo, la sombra estabiliza a los represores JAZ, aumentando los niveles de estos represores, lo que contribuye a reducir la capacidad de activación de defensas de la planta por los factores MYC.
- Chico JM, Fernández-Barbero G, Chini A, Fernández-Calvo P, Díez-Díaz M, Solano R. Repression of jasmonate-dependent defenses by shade involves differential regulation of protein stability of MYC transcription factors and their JAZ repressors in Arabidopsis. Plant Cell. 2014; pii: tpc.114.125047.
1er Meeting de la Iniciativa en NanoBiomedicina del CNB
 El viernes 10 de octubre de 2014 se celebrará en el Centro Nacional de Biotecnología del CSIC el 1er Meeting de la Iniciativa en NanoBiomedicina del CNB con el fin de dar visibilidad a la investigación que llevamos a cabo en el área de la NanoBiomedicina tanto grupos del CNB como grupos colaboradores de esta iniciativa pertenecientes al Campus de Excelencia UAM+CSIC.
El viernes 10 de octubre de 2014 se celebrará en el Centro Nacional de Biotecnología del CSIC el 1er Meeting de la Iniciativa en NanoBiomedicina del CNB con el fin de dar visibilidad a la investigación que llevamos a cabo en el área de la NanoBiomedicina tanto grupos del CNB como grupos colaboradores de esta iniciativa pertenecientes al Campus de Excelencia UAM+CSIC.
PROGRAMA
Nanomedicine (Chairs: Luis Ángel Fernández and Domingo F. Barber)
- 9:30 - María del Puerto Morales (Nanocrystals and Chemistry Group, Department of Biomaterials and Bioinspired Materials, ICMM, CSIC). Nanometrology, Standardization Methods for the synthesis and characterization of Magnetic Nanoparticles with applications in biomedicine
- 9:55 - Andrés de la Escosura (Nanoscience and Molecular Materials research Group, Department of Organic Chemistry, Faculty of Science, UAM). Hierarchical and Cooperative Supramolecular Strategies Towards Functional. Virus-Like Nanoparticles
- 10:20 - Luis Ángel Fernández (Department of Microbial Biotechnology & NanoBiomedicine Initiative, CNB, CSIC). Nanobodies and E. coli nanocells
- 10:45 - COFFEE
- 11:15 - Domingo F. Barber (Department of Immunology and Oncology & NanoBiomedicine Initiative, CNB, CSIC). Nanoparticles for targeted release in Immunotherapy
- 11:40 - Fernando Herranz (Advanced Imaging Unit, CNIC). Radionuclide-Fe3O4 nanoparticles: a core-doped nanosystem for dual hot spot imaging
- 12:05 - Aitziber López-Cortajarena / Alvaro Somoza (CNB-IMDEA Nanociencia Associated Unit). Nanoparticle uses in nanomedicine: Nanoparticule Modification and Gold nanoparticles as gene sensors
- 12:30 - Jesús M. de la Fuente (Instituto de Ciencia de materiales, ICMA-CSIC). Designing Inorganic Nanoparticles for Therapy and Diagnosis
- 13:15 - LUNCH
Single Molecule & Nanomachines (Chairs: José L. Carrascosa and Fernando Moreno Herrero)
- 14:30 - Pedro de Pablo (Departamento de Física de la Materia Condensada, UAM). Mechanical properties of viral particles
- 14:55 - Mauricio García Mateu (Centro de Biología Molecular Severo Ochoa, CSIC-UAM). Engineering at the nanoscale
- 15:25 - Julio Gómez-Herrero (Departamento de Física de la Materia Condensada, UAM). New AFM perspectives and modes
- 15:50 - Ricardo Arias-González (CNB-IMDEA Nanociencia Associated Unit). Laser manipulation of biomolecules and functional nanoparticles
- 16:15 - COFFEE
- 16:40 - Fernando Moreno-Herrero (Department of Macromolecular Structures & NanoBiomedicine Initiative, CNB, CSIC). DNA metabolism machineries at the single molecule level
- 17:05 - Borja Ibarra (CNB-IMDEA Nanociencia Associated Unit). Nanomotors at the nanoscale
- 17:30 - Luis Alberto Campos (Department of Macromolecular Structures, CNB, CSIC). Nanobiotechnology
Nueva iniciativa europea sobre experimentación con animales de granja
La semana pasada, la red intergubernamental European Cooperation in Science and Technology presentó SALAAM, una iniciativa para compartir los avances obtenidos en la ingeniería genética y el fenotipado de los animales de granja utilizados para experimentación. Esta acción, dirigida por el científico alemán Eckhard Wolf y la francesa Pascale Chavatte-Palmer, tiene como meta el desarrollo de modelos de laboratorio que puedan ser utilizados en medicina traslacional. Entre otros científicos europeos, el investigador del Centro Nacional de Biotecnología del CSIC (CNB) Lluís Montoliu participa como experto en la construcción de transgenes.
 Los pequeños animales de laboratorio, como los ratones, las ratas o las moscas de la fruta, han sido de una ayuda tremenda a la hora de estudiar y comprender los sistemas biológicos complejos. Sin embargo, para poder llevar a cabo futuros ensayos preclínicos más eficientes que los actuales parece que es necesario emplear conejos, cerdos, cabras, ovejas y vacas. Por eso, esta iniciativa se centra en los animales de granja como modelos para estudiar las enfermedades humanas.
Los pequeños animales de laboratorio, como los ratones, las ratas o las moscas de la fruta, han sido de una ayuda tremenda a la hora de estudiar y comprender los sistemas biológicos complejos. Sin embargo, para poder llevar a cabo futuros ensayos preclínicos más eficientes que los actuales parece que es necesario emplear conejos, cerdos, cabras, ovejas y vacas. Por eso, esta iniciativa se centra en los animales de granja como modelos para estudiar las enfermedades humanas.
Durante el tiempo que dure esta acción, Montoliu explica que “se organizarán diversas conferencias y cursos, abiertos a cualquier investigador interesado en el tema”. Los seminarios abordarán las nuevas tecnologías disponibles, la creación de bases de datos, la elección del modelo animal adecuado para cada tipo de experimento y, como no podía ser de otra manera, aspectos de bioética y bienestar animal.
Descubren que los linfocitos T pueden matar bacterias
En un estudio dirigido por investigadores del Centro Nacional de Biotecnología del CSIC (CNB) han descubierto que los linfocitos T son capaces de capturar por transinfección bacterias de las células dendríticas y matarlas. Estos sorprendentes resultados muestran que los linfocitos T, ejemplo clásico de inmunidad adaptativa, desarrollan funciones que hasta ahora se suponían exclusivas del sistema inmune innato.
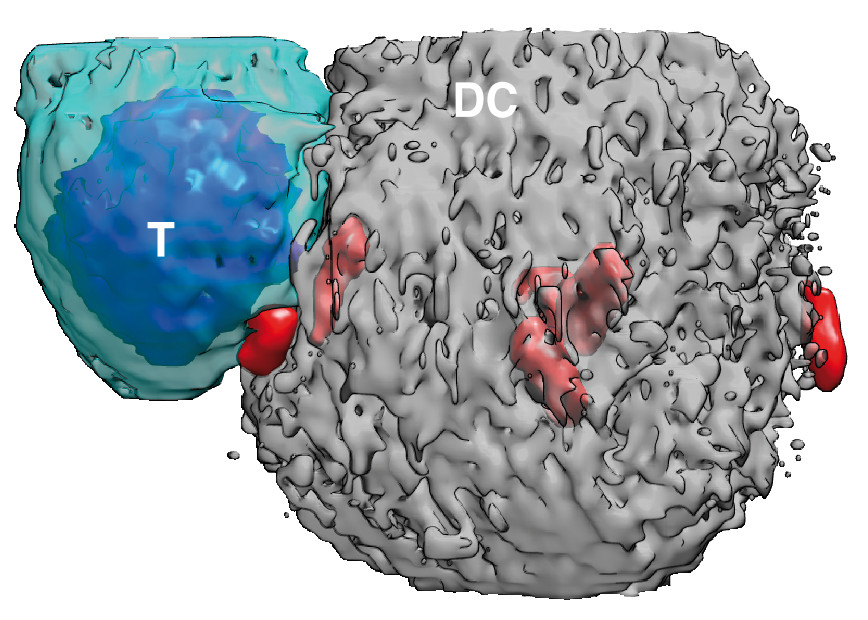 Los datos publicados en la revista Cell Host & Microbe "muestran por primera vez que los linfocitos T son capaces de capturar bacterias y matarlas", explica Esteban Veiga, investigador del CNB responsable principal de este estudio. Inesperadamente han descubierto que los linfocitos T transinfectados matan las bacterias de forma más eficiente que las propias células dendríticas.
Los datos publicados en la revista Cell Host & Microbe "muestran por primera vez que los linfocitos T son capaces de capturar bacterias y matarlas", explica Esteban Veiga, investigador del CNB responsable principal de este estudio. Inesperadamente han descubierto que los linfocitos T transinfectados matan las bacterias de forma más eficiente que las propias células dendríticas.
Analizando en su laboratorio del Instituto de Investigación Sanitaria Princesa (Hospital de Santa Cristina) las diferencias entre los linfocitos T, Aránzazu Cruz-Adalia observó que cuando son transinfectados, producen más citoquinas inflamatorias (como IL-6, TNFα e IFN-γ) que los no infectados. Además, son capaces de proteger in vivo de las infecciones bacterianas.
- Cruz-Adalia A, Ramirez-Santiago G, Calabia-Linares C, Torres-Torresano M, Feo L, Galán-Díez M, Fernández-Ruiz E, Pereiro E, Guttmann P, Chiappi M, Schneider G, Carrascosa JL, Chichón FJ, Martínez del Hoyo G, Sánchez-Madrid F, Veiga E. T cells kill bacteria captured by transinfection from dendritic cells and confer protection in mice. Cell Host Microbe. 2014; 15: 611-622.
Identifican relaciones evolutivas desconocidas entre virus distantes
Resolviendo la estructura tridimensional de proteínas de la cápsida vírica mediante criomicroscopía electrónica, investigadores del Centro Nacional de Biotecnología del CSIC (CNB) han identificado relaciones evolutivas hasta ahora desconocidas entre virus filogenéticamente distantes.
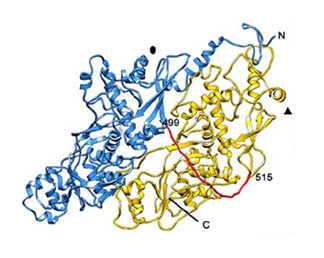 “Los virus evolucionan tan rápido que el análisis de sus secuencias genómicas no puede ser utilizado para encontrar relaciones entre virus filogenéticamente muy distantes”, explica José R. Castón, investigador responsable de un estudio que se acaba de publicar en la revista Proceedings of the National Academy of Sciences USA. En este trabajo presentan la estructura de la proteína de la cápsida del virus de Penicillium chrysogenum (PcV) y muestran el plegamiento de la proteína progenitora del linaje de los virus ARN de doble cadena (dsRNA).
“Los virus evolucionan tan rápido que el análisis de sus secuencias genómicas no puede ser utilizado para encontrar relaciones entre virus filogenéticamente muy distantes”, explica José R. Castón, investigador responsable de un estudio que se acaba de publicar en la revista Proceedings of the National Academy of Sciences USA. En este trabajo presentan la estructura de la proteína de la cápsida del virus de Penicillium chrysogenum (PcV) y muestran el plegamiento de la proteína progenitora del linaje de los virus ARN de doble cadena (dsRNA).
En colaboración con los grupos de Nùria Verdaguer (IBMB/CSIC), Niko Grigorieff (Brandeis University, USA) y Said Ghabrial (Kentuky University, USA), el equipo de Castón ha observado que la proteína de la cápside de este virus tiene dos mitades que, sin ser genéticamente similares, son dos α-hélices repetidas, indicando que provienen de la duplicación de un mismo gen. Este núcleo helicoidal conservado aumentó su complejidad por la duplicación inicial del gen y, seguidamente, por la inserción en puntos específicos (hotspots) de distintos segmentos peptídicos.
La identificación de los motivos estructurales ancestrales en el tipo de plegamiento conservado en el linaje de los virus dsRNA ha permitido la identificación de relaciones filogenéticas hasta ahora desconocidas.
- Luque D, Gómez-Blanco J, Garriga D, Brilot AF, González JM, Havens WM, Carrascosa JL, Trus BL, Verdaguer N, Ghabrial SA, Castón JR. Cryo-EM near-atomic structure of a dsRNA fungal virus shows ancient structural motifs preserved in the dsRNA viral lineage. PNAS. 2014; doi: 10.1073/pnas.1404330111.
La proteína WIP es esencial en la comunicación entre neuronas
Un estudio realizado por investigadores de CIBERNED del Centro Nacional de Biotecnología (CNB) y del Centro de Biología Molecular “Severo Ochoa” (CBMSO) ha identificado que la ausencia de la proteína WIP favorece la formación de contactos neuronales más grandes pero menos plásticos en respuesta a estímulos.
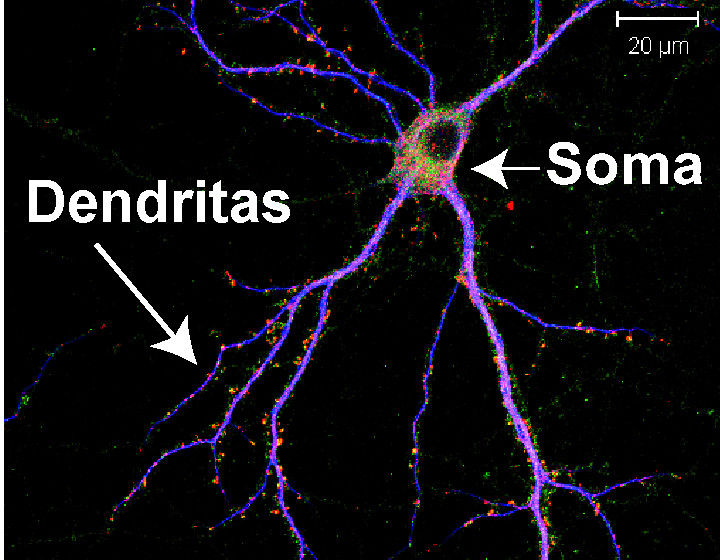 Estos contactos neuronales, conocidos como espinas dendríticas, posibilitan la conexión entre neuronas emisoras y receptoras, en estructuras conocidas como sinapsis, que cambian en número, tamaño y sensibilidad en respuesta a estímulos (plasticidad). Esta plasticidad sináptica es la base celular del aprendizaje y la memoria, relacionándose las alteraciones en la sinapsis con diferentes disfunciones neurológicas como el autismo, la esquizofrenia, la enfermedad de Alzheimer, el Síndrome de Down, la depresión o el trastorno bipolar.
Estos contactos neuronales, conocidos como espinas dendríticas, posibilitan la conexión entre neuronas emisoras y receptoras, en estructuras conocidas como sinapsis, que cambian en número, tamaño y sensibilidad en respuesta a estímulos (plasticidad). Esta plasticidad sináptica es la base celular del aprendizaje y la memoria, relacionándose las alteraciones en la sinapsis con diferentes disfunciones neurológicas como el autismo, la esquizofrenia, la enfermedad de Alzheimer, el Síndrome de Down, la depresión o el trastorno bipolar.
La colaboración entre los laboratorios dirigidos por Inés Antón (CNB-CSIC; CIBERNED) y Lola Ledesma (CBMSO), ambos situados en el Campus de Excelencia UAM-CSIC, aborda la influencia en estos procesos de la proteína WIP, aportando nuevos datos sobre el mecanismo que regula la morfología y la actividad de las espinas dendríticas, al haber permitido identificar dicha proteína como el punto de conexión entre la composición lipídica de la membrana y el citoesqueleto en estas estructuras. Así, se ha llegado a la conclusión de que la ausencia de WIP reduce los niveles del lípido esfingomielina en la membrana plasmática, activando así un conjunto de proteínas que aumentan la cantidad de filamentos de actina del esqueleto celular y favoreciendo la formación de espinas dendríticas más grandes y estables.
Al relacionar este descubrimiento con los datos de otros estudios que sugieren que la estabilidad de las espinas dendríticas se relaciona con un incremento en la memoria frente a estímulos concretos y con una disminución en la capacidad de aprendizaje, se ha conseguido identificar en un modelo de ratón la contribución de la proteína WIP al correcto funcionamiento sináptico, y en su aplicación a humanos podrían explicar el origen de las alteraciones neurológicas descritas en pacientes con modificaciones en la región génica que codifica esta proteína.
Según explica Ana Franco-Villanueva, coautora del estudio, “con la simple adición del lípido esfingomielina hemos corregido el defecto sináptico en un modelo de cultivo neuronal de ratón deficiente en WIP, proporcionando la primera estrategia para el futuro tratamiento de los pacientes”.
- Franco-Villanueva A, Fernández-López E, Gabandé-Rodríguez E, Bañón-Rodríguez I, Esteban JA, Antón IM, Ledesma MD. WIP modulates dendritic spine actin cytoskeleton by transcriptional control of lipid metabolic enzymes. Hum Mol Genet. 2014; doi: 10.1093/hmg/ddu155.
Genome editing: A new tool for generating genetically modified animal models
El 27 de mayo de 2014 se celebra en el Centro Nacional de Biotecnología del CSIC una jornada sobre edición genómica.
Organizada en por el Servicio de Transgénesis CNB-CBMSO y Sigma, quien patrocina el evento, se llevará a cabo en el Salón de Actos del CNB desde las 10:20 hasta las 14:30. La inscripción se realiza a través del formulario.
Programa:
- Silvia Di Meglio (Sigma-Aldrich, EMEA)
Using CRISPr and ZFN Technologies for creation of Transgenic animals - Belen Pintado (CNB-CBMSO, Madrid)
Genome editing. Impact on animal use - Roger Badia (Hospital Germans Trias i Pujol)
Working with ZFN: Practical issues learnt from HIV research - Ignacio Anegon (INSERM UMR, Nantes)
Gene editing of the rat genome using ZFNs and other gene-specific nucleases - Davide
Seruggia (CNB-CSIC, Madrid)
From TALENs to CRISPR: new tools for genome editing

La proteína de la envoltura del coronavirus del SARS determina su virulencia
En un estudio que investigadores del Centro Nacional de Biotecnología del CSIC (CNB) han publicado en la revista PLoS Pathogens se identifica la proteína viral clave para que el coronavirus del SARS produzca lesiones pulmonares.
 Luis Enjuanes, el investigador del CNB responsable principal de este trabajo, explica que “el coronavirus del SARS induce una respuesta inflamatoria incontrolada que causa daño pulmonar, edema, hipoxemia severa y eventualmente la muerte. Lo que hemos encontrado es que la actividad de la proteína E como transportadora de iones promueve las lesiones que causa el SARS”. Estos descubrimientos podrían llegar a usarse para desarrollar una terapia contra esta enfermedad.
Luis Enjuanes, el investigador del CNB responsable principal de este trabajo, explica que “el coronavirus del SARS induce una respuesta inflamatoria incontrolada que causa daño pulmonar, edema, hipoxemia severa y eventualmente la muerte. Lo que hemos encontrado es que la actividad de la proteína E como transportadora de iones promueve las lesiones que causa el SARS”. Estos descubrimientos podrían llegar a usarse para desarrollar una terapia contra esta enfermedad.
En colaboración con científicos de la Universitat Jaume I de Valencia, el investigador del CNB José Luis Nieto Torres ha generado coronavirus del SARS en los que su proteína de la envoltura carece de sus propiedad como transportadora de iones. Sin esta función, el virus se atenúa, reduciéndose la respuesta inflamatoria dañina que provoca a los ratones infectados.
Como hay varios virus muy patógenos con proteínas transportadoras de iones similares a las de estos coronavirus, Enjuanes comenta que sus resultados podrían ser relevantes en el estudio de infecciones de virus como el de la gripe A, el sida o la hepatitis C.
- Nieto-Torres JL, Dediego ML, Verdiá-Báguena C, Jimenez-Guardeño JM, Regla-Nava JA, Fernandez-Delgado R, Castaño-Rodriguez C, Alcaraz A, Torres J, Aguilella VM, Enjuanes L. Severe Acute Respiratory Syndrome Coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis. PLoS Pathog. 2014; 10(5): e1004077.
Descubren que la proteína Vps4 protege al genoma viral
Los virus ARN, entre los que se encuentran muchos patógenos para humanos, animales y plantas, replican sus genomas en membranas del interior de las células. En estas membranas los virus reclutan factores celulares que participan en el ensamblaje y actividades de los complejos replicativos. Estos agregados macromoleculares, que suelen ensamblarse en vesículas con aperturas al citosol o “esférulas”, se encargan de fabricar múltiples copias del genoma viral que se incorporarán posteriormente en las nuevas partículas virales infectivas. Aunque se han identificado numerosos factores necesarios para el ensamblaje funcional de los orgánulos de replicación viral se desconoce la función exacta de la mayoría de ellos.
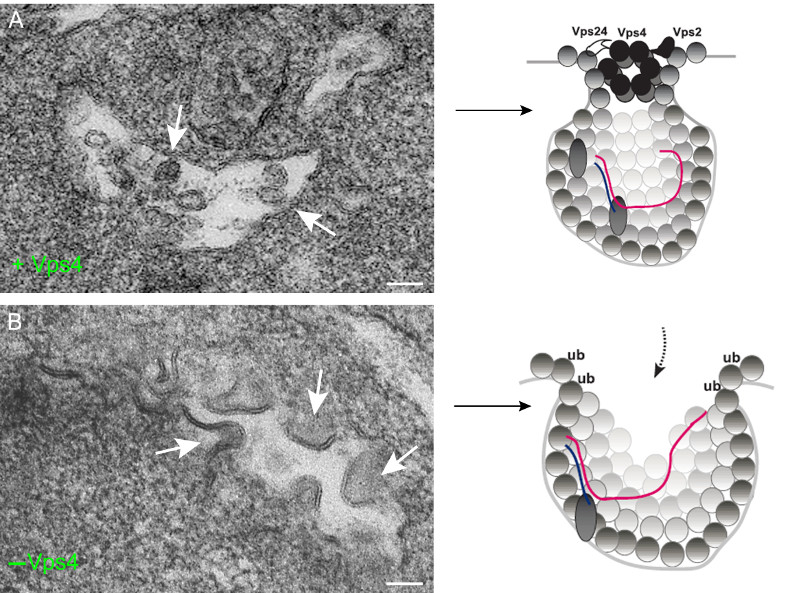 En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
Vps4 es un componente de las proteínas celulares ESCRT y de la familia de las ATPasas AAA+ que usan ATP para remodelar estructuras macromoleculares en las células. Vps4 participa en una variedad de procesos biológicos como por ejemplo la fusión de membranas para la formación de vesículas. Utilizando nuevas técnicas de imagen en microscopía electrónica y según acaban de publicar en la revista PLoS Pathogens, los autores han demostrado que Vps4 participa en la estabilización del poro o cuello de las esférulas a través del cual se produce el intercambio de materiales con el citosol. Esta apertura controlada es necesaria para que el complejo replicativo desempeñe sus funciones y para que el ARN viral quede protegido de la degradación por nucleasas.
A diferencia de lo que ocurre en los procesos celulares habituales en los que participa, Vps4 se incorpora de manera estable en las vesículas virales o esférulas y pasa a formar parte del complejo replicativo. De hecho cuando Vps4 no está presente se forman esférulas totalmente abiertas, sin constricción o cuello y en las que el ARN viral de nueva síntesis está desprotegido. Los autores proponen que Vps4 y otras proteínas ESCRT son indispensables para la deformación de las membranas y el ensamblaje de los complejos replicativos de Tombusvirus. Es muy probable que otros virus de plantas y animales que transforman membranas celulares para construir esférulas también utilicen estas proteínas para ensamblar sus replicasas y proteger sus genomas recién sintetizados.
- Barajas D, Martín IFdC, Pogany J, Risco C, Nagy PD. Noncanonical role for the host Vps4 AAA+ ATPase ESCRT protein in the formation of Tomato bushy stunt virus replicase. PLoS Pathog. 2014; 10(4): e1004087.
