
Descifrado el mecanismo molecular de una enfermedad rara que causa inmunodeficiencias graves
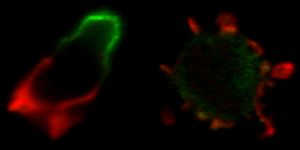
- Se trata del síndrome de WHIM, una enfermedad producida por mutaciones en un receptor de la membrana celular
- Un nuevo estudio liderado por investigadores del CSIC identifica dianas terapéuticas para el desarrollo de compuestos que permitan una respuesta inmunitaria normal
El síndrome de WHIM es una enfermedad congénita que causa una inmunodeficiencia severa y el aumento de infecciones, tanto bacterianas como víricas. Este síndrome está causado por mutaciones en el gen CXCR4, un receptor implicado en la adhesión y migración celular, entre otras funciones, pero hasta ahora se desconocía cómo estas mutaciones afectaban a su mecanismo de acción.
Investigadores del CSIC, en colaboración con diferentes grupos científicos nacionales y europeos, han identificado el mecanismo molecular responsable de esta enfermedad. El trabajo, publicado en la revista Proceedings of the National Academy of Sciences USA (PNAS), combina técnicas de imagen y análisis bioinformático y demuestra que las mutaciones en CXCR4 afectan a su dinámica y organización en la membrana celular, lo que afecta a la migración celular.
Todo al verde: el poder de las plantas como una herramienta para cuidar el entorno

- Investigadores del CSIC estudian la capacidad de las plantas para eliminar contaminantes del suelo y las aguas residuales
- Desde el Centro Nacional de Biotecnología (CNB-CSIC) estudian la 'lemna', la lenteja acuática que captura el arsénico del suelo
La ropa, los champús, la colonia, cientos de pinturas, medicinas, neumáticos, todo tipo de alimentos, un curioso cobijo para pequeños individuos, el oxígeno, la vida… Todo esto y más nos proporcionan las plantas. Su papel en la biodiversidad es incuestionable; son las ingenieras del medioambiente, base de todas las cadenas tróficas y un seguro de vida para capturar y almacenar el carbono.
En el planeta existen más de 320.000 especies distintas de plantas, con diferentes formas y colores. Su existencia en sí supone un reto a la creatividad, pero además son una herramienta que puede resultar muy útil para mantener el entorno libre de compuestos tóxicos, metales pesados o plaguicidas. En el Centro Nacional de Biotecnología (CNB-CSIC), el grupo de Antonio Leyva trata de sacar el máximo partido a la Lemna, una especie capaz de “atrapar” el arsénico. Para lograrlo necesitan comprender primero qué genes concretos están involucrados en ese proceso capaz de absorber, descomponer y eliminar aquello que se desee.
Revelan la estructura y el mecanismo de la alfa2-macroglobulina, una molécula que retira enzimas “caducadas” del plasma sanguíneo
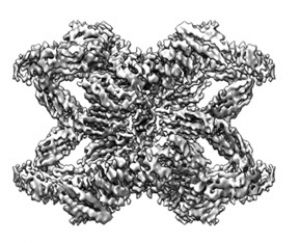
- La alfa2-macroglobulina humana participa en la eliminación de enzimas del plasma sanguíneo que ya han cumplido su función y no son necesarias
- Su mecanismo de acción, poco habitual, afecta a muchas funciones fisiológicas y los investigadores lo comparan con una trampa de planta carnívora
Un trabajo conjunto del Instituto de Biología Molecular de Barcelona (IBMB) y del Centro Nacional de Biotecnología (CNB), ambos del Consejo Superior de Investigaciones Científicas (CSIC), detalla a nivel molecular el mecanismo que utiliza la molécula alfa2-macroglobulina humana (hA2M) para inhibir muchos tipos de endopeptidasas, unas enzimas que cortan otras proteínas y péptidos. Este peculiar mecanismo permite la inhibición de diferentes clases de endopeptidasas y los investigadores lo comparan de forma gráfica con una trampa de planta carnívora.
Los resultados, publicados en la revista Proceedings of the National Academy of Sciences (PNAS), son el resultado de nueve años de trabajo con técnicas avanzadas de criomicroscopía electrónica por parte de los equipos liderados por F. Xavier Gomis-Rüth, del IBMB-CSIC, y José R. Castón, del CNB-CSIC, en colaboración con la Unidad de Microscopía Electrónica del Instituto de Salud Carlos III (ISCIII) y la Universidad de Leeds (Inglaterra).
Jornada de Búsqueda de Sinergias entre el Instituto de Química Médica y el Centro Nacional de Biotecnología

Hoy se ha celebrado en el CNB la Jornada "Búsqueda de Sinergias entre el Instituto de Química Médica y el Centro Nacional de Biotecnología" con la participación de personal investigador de ambos centros.
La jornada, en la que han participado representantes de 11 grupos del IQM y 26 grupos del CNB, ha servido como punto de contacto para conocer las posibilidades de colaboración entre los grupos de ambos centros. En ella se ha hablado de temas relacionados con la química y la biomedicina desde el desarrollo de vertebrados hasta el diseño molecular, el uso de diferentes moléculas con propiedades farmacológicas, las estructuras de virus y complejos celulares así como mecanismos de respuesta inmune y procesos inflamatorios abarcando.
Hay que destacar las posibilidades y aplicaciones del cribado de compuestos con potencial terapéutico en los contextos de la plataforma de antivirales y de resistencia a antibióticos, al igual que la determinación de estructuras e interacciones moleculares a nivel atómico, todo con el objetivo último de avanzar en la One Health Initiative en sus tres aspectos de salud humana, animal y ambiental.
Fernando Corrales recibe el Premio “Juan Pablo Albar Proteome Pioneer Award ” de la Asociación Europea de Proteómica

Durante el XIV Congreso anual de la Asociación Europea de Proteómica (EuPA) celebrado en Leipzig (Alemania), el investigador del CNB-CSIC Fernando Corrales ha recibido el premio “Proteome Pioneer Award Juan Pablo Albar” por su trayectoria científica en el área de la proteómica.
Premio Juan Pablo Albar a los pioneros de la proteómica
Con este premio, el objetivo de la EuPA es honrar a una persona con una trayectoria de excelencia en el ámbito de la investigación, que ha contribuido al establecimiento de los principios básicos sobre el estudio de las proteínas a gran escala y al desarrollo de la Proteómica en Europa. El premio Juan Pablo Albar recuerda la memoria de uno de los pioneros en esta área científica, también investigador del CNB-CSIC.
Trayectoria científica
Fernando Corrales dirige el Laboratorio de Proteómica del CNB-CSIC. Sus investigaciones se centran en el estudio de los mecanismos asociados a la función y enfermedad hepática, utilizando enfoques de proteómica y genómica combinados en una estrategia basada en la biología de sistemas. Su actividad investigadora ha dado lugar a la publicación de más de 180 publicaciones científicas y es miembro del consejo editorial de diferentes revistas especializadas en Proteómica y Hepatología. Ha sido el Coordinador General de la Plataforma de Recursos Moleculares y Bioinformáticos (PRB3) y de ProteoRed-ISCIII (Red Española de Proteómica). Es miembro de los Consejos Ejecutivos de la Sociedad Española de Proteómica (SEProt), de la Asociación Europea de Proteómica (EuPA). Dentro del proyecto HUPO, lidera la iniciativa centrada en hígado y los Proyectos BD y C-HPP.
Víctor de Lorenzo, investido doctor honoris causa por la Denmark Technical University

El investigador del CNB Víctor de Lorenzo recibió el pasado día 29 de abril de 2022 un doctorado honoris causa de la Denmark Technical University (DTU) en una ceremonia que tuvo lugar en el campus de Lyngby. En la ceremonia actuaron como anfitriones la presidenta de la Junta Directiva de la DTU, Karin Markides, el vicerrector Anders Bjarklev, el prorrector Rasmus Larsen y el director de la Asociación Politécnica (FP), Andreas Ipsen.
En el evento se celebraron los nuevos doctorados de la universidad y se concedieron dos doctorados honoris causa a investigadores de otra universidad con una larga y estrecha relación con la DTU: Sang Yup Lee, investigador y vicepresidente del Instituto Avanzado de Ciencia y Tecnología de Corea (KAIST) por sus contribuciones en la ingeniería de sistemas y Víctor de Lorenzo, profesor de investigación del CSIC en el CNB por sus actividades pioneras en biología molecular, su contribución a la biotecnología y la microbiología ambiental.
Un equipo del CNB-CSIC produce anticuerpos neutralizantes eficaces contra la covid-19
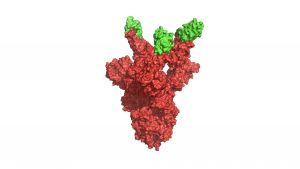
- Estos anticuerpos, basados en el sistema inmunitario de los dromedarios, tienen potencial terapéutico en pacientes inmunodeficientes o no protegidos por las vacunas
- El estudio ha demostrado en modelos animales que son activos frente a las variantes más virulentas de SARS-CoV-2 y que pueden proteger hasta el 100% de los infectados
Investigadores del CSIC han obtenido anticuerpos neutralizantes eficaces frente a las variantes más virulentas del SARS-CoV-2 y que pueden utilizarse como terapia en pacientes de covid-19. Los investigadores, que han publicado el estudio en Frontiers in Immunology, han producido los anticuerpos mediante cultivos celulares en el laboratorio y afirman que la producción ya puede escalarse para su aplicación clínica. Además, estos anticuerpos tienen un gran potencial para la detección del virus, por lo que pueden ser de gran utilidad para diferentes formatos de test antigénicos del SARS-CoV-2. El CSIC ha patentado esta tecnología y busca empresas interesadas en llevar estos anticuerpos al mercado.
Los investigadores del Centro Nacional de Biotecnología (CNB-CSIC) dirigidos por Luis Ángel Fernández y José María Casasnovas seleccionaron los nanoanticuerpos que mejor se unían a la región de la proteína viral S (spike) del SARS-CoV-2 y que bloqueaban la entrada del virus en la célula. Los ensayos in vitro en células infectadas con SARS-CoV-2 identificaron aquellos con una mayor actividad neutralizante del virus en la plataforma de antivirales del instituto del CSIC, dirigida por los investigadores Urtzi Garaigorta y Pablo Gastaminza.
Descubren una nueva función bacteriana para sobrevivir al sistema inmune
- Investigadores del Centro Nacional de Biotecnología desvelan la doble función del gen comk del patógeno Staphylococcus aureus para reparar su ADN y modificar su metabolismo durante la infección
- Este trabajo permitirá investigar nuevas dianas terapéuticas para reforzar el sistema inmunitario frente a infecciones hospitalarias
Un equipo internacional liderado por el Centro Nacional de Biotecnología (CNB-CSIC) describe en Nature Communications una nueva función de genes del patógeno Staphylococcus aureus que le permite sobrevivir al sistema inmune del hospedador. Este hallazgo abre la puerta al desarrollo de dianas terapéuticas que permitan reforzar el sistema inmune frente a infecciones hospitalarias.
Las bacterias presentan una propiedad llamada competencia natural. Esta se basa en la acción de un complejo proteico de su membrana que puede transportar ADN externo -de la célula que infectan o de otras bacterias cercanas- al interior de la bacteria e incorporarlo al suyo propio. Mediante dicho mecanismo, la bacteria modifica su propia información genética. “Es una manera que tienen de evolucionar”, apunta Daniel López, jefe del grupo Biología Molecular de las Infecciones en el CNB y quien ha dirigido la investigación.
Sin embargo, quedaba por resolver una incógnita relacionada con la información genética responsable de semejante función. “Hay muchas especies bacterianas que, aunque tienen los genes para producir esa maquinaria, nunca se ha visto que sean capaces de adquirir genes de fuera. Una cuestión que ha estado sobre la mesa durante décadas es: ¿cómo es posible que los genes de competencia natural estén en bacterias competentes?”, señala López.
Descubiertas nuevas funciones de la proteína reparadora humana HELB

- HELB es una helicasa implicada en importantes procesos de replicación y reparación del genoma
- Investigadores del CSIC utilizan la tecnología de pinzas ópticas para observar el comportamiento de esta proteína sobre la molécula de ADN
Sobre el genoma de cada célula actúan numerosas proteínas que leen, duplican o reparan el ADN. Para que estas proteínas puedan actuar es necesario abrir la doble hélice del ADN, un paso esencial en los procesos de replicación o reparación de esta molécula. Las helicasas son pequeñas máquinas moleculares que realizan esta acción. Pese a su importancia, existen múltiples helicasas cuya función concreta está aún por identificar.
Ahora, un trabajo publicado en Proceedings of the National Academy of Sciences (PNAS) describe la importancia de la proteína humana HELB. Investigadores del Consejo Superior de Investigaciones Científicas (CSIC) en colaboración con la Universidad de Bristol han observado que HELB, una helicasa hasta ahora poco conocida pero cuyas mutaciones se asocian a diversos problemas de salud y al desarrollo de tumores, parece cumplir un papel fundamental en la reparación del ADN.

