Un estudio evolutivo abre la puerta a una estrategia eficaz frente a bacterias resistentes a los antibióticos
- La adquisición de mutaciones de resistencia a antibióticos puede dar lugar a la sensibilización frente a otros antibióticos, un fenómeno conocido como sensibilidad colateral
- Comprender la evolución de la resistencia a los antibióticos podría facilitar el diseño de nuevas estrategias terapéuticas utilizando antibióticos de uso habitual
La resistencia de las bacterias a los antibióticos es, tal como indican la OMS y la ONU, uno de los mayores problemas actuales de salud pública. Dicha resistencia hace más difícil -en ocasiones imposible- tratar las infecciones bacterianas, así como desarrollar prácticas clínicas como trasplantes o tratamientos anticancerígenos que requieren un control de posibles infecciones. Entre estas, tienen gran relevancia las causadas por Pseudomonas aeruginosa, uno de los patógenos oportunistas que con mayor frecuencia produce infecciones en pacientes hospitalizados o con patologías previas, incluyendo inmunodeficiencias y fibrosis quística. Esta bacteria tiene una baja sensibilidad a los antibióticos, y una gran capacidad para adquirir mutaciones de resistencia a los mismos, de ahí el interés en encontrar terapias eficaces contra ella. En este contexto, además de desarrollar nuevos antibióticos, algo económicamente costoso, es necesario hacer un mejor uso de los que ya tenemos.
Un nuevo estudio de los investigadores del CSIC Sara Hernando-Amado, Pablo Laborda, José Ramón Valverde y José Luis Martínez publicado en la revista Proceedings of the National Academy of Sciences (PNAS), da un paso más en la búsqueda de alternativas terapéuticas basadas en el uso de antibióticos ya existentes. Analizando la evolución de diferentes mutantes de P. aeruginosa en presencia de distintos antibióticos, han identificado un patrón conservado de sensibilidad frente a otros antibióticos (un fenómeno llamado sensibilidad colateral) que podría tener aplicación clínica.
Una combinación de métodos bioquímicos y estructurales desvela el mecanismo de acción antibacteriano de la enzima de origen viral Pae87
Estimaciones recientes indican que las bacterias resistentes a los antibióticos ya causan más de un millón de muertes al año en todo el mundo, lo que hace necesario la búsqueda de tratamientos alternativos. En este sentido, los virus bacterianos o bacteriófagos que infectan bacterias, se convierten en un prometedor objeto de investigación. Estos bacteriófagos (o fagos para abreviar) matan a sus huéspedes al producir un tipo de enzimas (endolisinas) que degradan la capa de peptidoglicano en la pared celular bacteriana. Pero el uso de las endolisinas para erradicar bacterias aún requiere una mejor comprensión de su estructura y función.
Un reciente estudio de colaboración publicado en la revista Acta Crystallographica section D describe la caracterización estructural y funcional de la enzima endolisina Pae87 del fago JG004 de Pseudomonas aeruginosa. Investigadores de los laboratorios de Pedro García en el Centro de Investigaciones Biológicas Margarita Salas (CIB-CSIC) y Mark van Raaij en el Centro Nacional de Biotecnología (CNB-CSIC) han llevado a cabo estudios bioquímicos y estructurales que han permitido resolver la estructura de la proteína y su función.
Identificado un gen que regula la localización subterránea de los tubérculos de patata

- Si las condiciones ambientales son buenas, las plantas de patata utilizan los azucares en la parte aérea para crecer, mientras que para sobrevivir al invierno los acumulan en tubérculos bajo tierra en forma de almidón
- El gen BRANCHED1b, que regula varios procesos moleculares relacionados con periodos de inactividad vegetal, es también responsable de que los tubérculos se formen bajo tierra
Investigadores del Consejo Superior de Investigaciones Científicas identifican una nueva función del gen BRANCHED1b, que ha resultado ser esencial para la formación de tubérculos bajo tierra. El trabajo, que se publica ahora en Nature Plants muestra que mutaciones en este gen afectan a la capacidad de distinguir entre tallos aéreos y subterráneos (llamados estolones) en las plantas de patata. De esta manera en las plantas mutantes se produce una distribución errónea de los azucares y de las señales que controlan la formación de tubérculos y estas desarrollan tubérculos aéreos, expuestos a las inclemencias del tiempo y los animales.
El equipo del CSIC dirigido por Pilar Cubas, en colaboración con el de Salomé Prat, en el Centro Nacional de Biotecnología (CNB-CSIC) ha identificado que el gen llamado BRANCHED1b (BRC1b) es esencial para que los tubérculos se formen únicamente en la parte subterránea de la planta. Cubas explica sus resultados “utilizando como modelo plantas de patata silvestres y mutantes que no expresaban este gen, estudiamos la formación de tallos aéreos y subterráneos (llamados estolones) durante seis semanas. Así observamos que los mutantes producían más tallos aéreos, pero menos estolones que las plantas silvestres. Pero el cambio más llamativo se refería a la formación de tubérculos: los mutantes producían muchos menos tubérculos subterráneos y de menor tamaño, y además, empezaron a producir tubérculos aéreos.”
Mutaciones de ómicron ya existían en pacientes infectados meses antes de ser dominantes
Identificada una posible estrategia terapéutica basada en la compensación fisiológica de la resistencia bacteriana a los antibióticos

- Las mutaciones que producen resistencia bacteriana pueden suponer un coste fisiológico que reduce su crecimiento cuando cesa el tratamiento antibiótico
- Esta desventaja puede servir para implementar tratamientos discontinuos utilizando antibióticos ya disponibles
Las resistencias bacterianas se deben, entre otras causas, a la adquisición de variaciones genéticas (por ejemplo, mutaciones) como consecuencia de la presión selectiva que ejercen los antibióticos. En algunas ocasiones, un incremento de resistencia a un antibiótico puede suponer para las bacterias un coste fisiológico (por ejemplo, un peor crecimiento) cuando crecen en ausencia del mismo, algo que podría explotarse clínicamente. En el caso de Pseudomonas aeruginosa, un patógeno oportunista que produce infecciones en pacientes hospitalizados o con patologías previas, tiene una elevada resistencia intrínseca a los antibióticos y una gran capacidad para adquirir mutaciones de resistencia a los mismos, de ahí la importancia de encontrar tratamientos eficaces.
Investigadores del Consejo Superior de Investigaciones Científicas (CSIC), usando ensayos de evolución experimental, han identificado una posible estrategia terapéutica que aprovecha el coste fisiológico asociado a la adquisición de resistencia bacteriana. El nuevo trabajo de Sara Hernando-Amado, Pablo Laborda, José Ramón Valverde y José Luis Martínez publicado en Molecular Biology and Evolution plantea un escenario en el que este coste de crecimiento podría favorecer una bajada, o incluso reversión, de la resistencia a los antibióticos. En estos casos, se podrían realizar tratamientos discontinuos de antibióticos ya que, pasado un breve periodo de tiempo, las bacterias volverían a ser sensibles al antibiótico inicial. El conocimiento de la evolución y el coste fisiológico asociado se convierte así en una herramienta más para la posible reutilización de antibióticos ya disponibles en clínica.
El grupo de Alvaro San Millán recibe el premio PRAN a la mejor publicación científica sobre resistencia a los antibióticos
El trabajo del grupo de Alvaro San Millán del CNB-CSIC ha sido galardonado con uno de los premios PRAN de la Agencia Española del Medicamento y Productos Sanitarios (AEMPS).
Su artículo "Permassive transmission of a carbopenem resistance plasmid in the gut microbiota of hospitalized patients" publicado en Nature Microbiology en el año 2021 ha sido considerado la mejor publicación científica sobre resistencia a antibuioticos del año 2021.
Un estudio muestra cómo el fármaco plitidepsina inhibe la reproducción del SARS-CoV-2 dentro de las células
- Actúa sobre las células e impide la formación de los espacios donde se reproduce el virus, según revela un estudio coliderado por investigadoras del CSIC e IrsiCaixa
Un estudio coliderado por investigadores del Consejo Superior de Investigaciones Científicas (CSIC) ha mostrado cómo actúa el fármaco aplidin (plitidepsina) para inhibir la replicación del coronavirus SARS-CoV-2 dentro de las células humanas: el fármaco impide la formación de compartimentos celulares donde se replica el material del virus. De este modo, interfiere en las células y evita la progresión de la infección viral. Su funcionamiento refuerza su potencial para combatir futuras variantes del SARS-CoV-2, pues este paso de la replicación del virus es común a todas las variantes del virus. El estudio, en fase de revisión, se presentó ayer en la Conferencia sobre Retrovirus e Infecciones Oportunistas (CROI), en Barcelona.
El fármaco plitidepsina, comercializada con el nombre de aplidin, ha sido uno de los fármacos que ha demostrado mayor eficacia para bloquear la replicación del SARS-CoV-2 en experimentos de laboratorio. “Cuando pusimos en marcha este estudio para analizar la actividad antiviral de plitidepsina en la célula, no esperábamos descubrir que su diana de actuación se encontraba en un estadio tan inicial de la replicación del SARS-CoV-2”, explica Cristina Risco, investigadora del Centro Nacional de Biotecnología (CNB-CSIC) y co-líder del estudio junto a investigadores del Instituto de Investigación del Sida IrsiCaixa, el Centro de Investigación en Sanidad Animal (IRTA-CReSA) y la farmacéutica PharmaMar.
El proyecto Rock-Eaters analizará cómo avanza la vida en lavas y rocas de áreas deglaciadas de la Antártida

- La expedición antártica será del 14 de febrero al 15 de marzo
- Los resultados ayudarán a analizar como contribuyen los microorganismos a la respuesta de los ecosistemas antárticos a los cambios ambientales
- Gracias a las herramientas que han desarrollado muchos de los análisis se hacen in situ, sin necesidad de llegar al laboratorio
Madrid, 10 de febrero de 2022. Cuando la lava de un volcán se enfría o por el retroceso glaciar quedan al descubierto suelos y rocas previamente cubiertas por hielo, los microorganismos son los primeros seres vivos que los colonizan. Esta colonización pionera es el paso previo e imprescindible para que se desarrollen otras formas de vida más complejas. Estas comunidades microbianas son las que se proponen estudiar en el proyecto ROCK-EATERS en el que participan Asunción de los Ríos y Fernando Garrido del Museo Nacional de Ciencias Naturales de Madrid (MNCN) y Javier Tamames del Centro Nacional de Biotecnología (CNB), ambos del CSIC. Los datos obtenidos les permitirán descubrir una diversidad antártica desconocida y caracterizar sus dinámicas para en el futuro poder valorar su resiliencia frente al cambio climático. El curso de la expedición se podrá seguir a través del blog Rock-eaters Antarctic Expedition: searching microbial life in rocks.
El recubrimiento de las nanopartículas magnéticas es clave para sus aplicaciones biomédicas
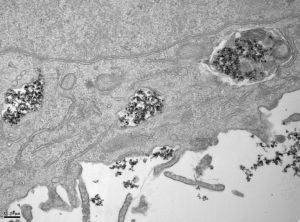
- Las nanopartículas de óxido de hierro muestran un gran potencial para numerosas aplicaciones biomédicas, especialmente para el tratamiento del cáncer
- Investigadores del CSIC identifican el recubrimiento de las nanopartículas de óxido de hierro como el principal factor que determina el tráfico y la velocidad de degradación intracelular
Madrid 4 febrero 2022. Las nanopartículas magnéticas de óxido de hierro han mostrado en la última década un alto potencial en medicina, habiéndose obtenido resultados prometedores en varias aplicaciones médicas. A pesar de estos resultados prometedores, para aprobar su uso clínico de manera generalizada es fundamental entender cómo se comportan en el interior de las células, qué rutas intracelulares activan, y cómo se degradan. Este comportamiento intracelular determinará en gran medida tanto los efectos terapéuticos de las nanopartículas como su posible toxicidad.
El nuevo trabajo de investigadores del Consejo Superior de Investigaciones Científicas CSIC liderados por Domingo F Barber y publicado en la revista Biomaterials, desvela nuevos aspectos del comportamiento intracelular de las nanopartículas. Como explica Barber, investigador en el Centro Nacional de Biotecnología (CNB-CSIC) “para su uso en aplicaciones biomédicas, las nanopartículas de óxido de hierro se recubren con diferentes tipos de moléculas o polímeros para hacerlas más biocompatibles, estables y evitar que formen agregados que podrían generar trombos. Según el tipo de recubrimiento utilizado, cuando las nanopartículas entran en contacto con el entorno biológico se producen diferentes interacciones con las proteínas del medio, afectando su tamaño final, la vía de captación celular, así como el tránsito que siguen hasta su degradación en la maquinaria celular que se encarga de este proceso (los endolisosomas)”.