
Redefinen el mecanismo y la relevancia del “patinaje” de polimerasas de virus ARN

- Las polimerasas son proteínas encargadas de copiar el material genético de los organismos, tanto ADN como ARN
- Los virus pequeños pueden optimizar la expresión de más genes utilizando el “patinaje de la polimerasa”, un mecanismo que permite producir dos proteínas a partir de un mismo gen
- Un equipo de investigación internacional identifica nuevos patrones en este funcionamiento de este mecanismo y su relevancia en la evolución viral
Un equipo internacional liderado por un grupo del Centro nacionald e Biotecnología en el Consejo Superior de Investigaciones Científicas (CSIC), detalla el mecanismo y la importancia de una estrategia de expresión génica llamada “patinaje” de la polimerasa en diversos virus cuyo genoma está formado por ARN (virus ARN). El trabajo, en colaboración con instituciones de España, Francia y China se publica en la revista mBio.
Algunas familias de virus ARN, generalmente con genomas de tamaño reducido, han desarrollado estrategias para maximizar la poca información genética que contienen. Una de ellas consiste en el “patinaje de la polimerasa” que, con la adición poco frecuente de un nucleótido extra al copiar el genoma viral, dan lugar a una proteína alternativa, o a una versión truncada de la proteína original, en ambos casos originando factores virales con funciones muy importantes durante la infección. Ejemplos de esto son la glicoproteína del virus Ébola, o P3N-PIPO en los virus vegetales de la familia Potyviridae.
Descubren un mecanismo que acelera la evolución de bacterias resistentes a los antibióticos

- Un equipo del CNB-CSIC desvela la capacidad de las bacterias resistentes a antibióticos de evolucionar de manera diferente gracias a las moléculas conocidas como plásmidos
- Este nuevo mecanismo molecular se ha observado tanto ‘in vitro’ como en la microbiota del intestino de pacientes hospitalizados
La resistencia de las bacterias a los antibióticos es una de las principales amenazas globales de salud pública. Según la Organización Mundial de la Salud (OMS), esta pandemia silenciosa causa más de un millón de muertes al año y podría convertirse en 2050 en la primera causa de mortalidad. En este contexto, un equipo del Centro Nacional de Biotecnología (CNB) del CSIC, dependiente del Ministerio de Ciencia, Innovación y Universidades (MICIU), desvela que las bacterias resistentes a antibióticos son capaces de evolucionar de manera diferente gracias a los plásmidos, unos elementos genéticos móviles responsables de que los mecanismos de resistencia se diseminen entre las bacterias.
Este trabajo, publicado en la revista Nature Ecology and Evolution y realizado en colaboración con el Instituto Ramón y Cajal de Investigación Sanitaria (IRYCIS) del Hospital Universitario Ramón y Cajal de Madrid, revela un nuevo mecanismo molecular de la evolución las bacterias multirresistentes que ocurre tanto in vitro como en la microbiota intestinal de pacientes hospitalizados, es decir, en el conjunto de microorganismos que habita en el intestino.
Identificado un nuevo factor clave en la tolerancia a arsénico en plantas
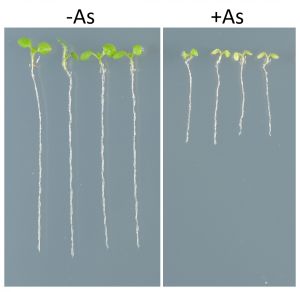
- La presencia de arsénico en los suelos reduce el desarrollo de las plantas y produce graves pérdidas económicas en la agricultura.
- Un equipo del CNB-CSIC identifica el factor de transcripción GLABRA2 como un nuevo regulador de la tolerancia al arsénico en plantas.
El arsénico, un elemento altamente tóxico presente de forma natural en el medio ambiente, constituye una amenaza global para los organismos vivos, comprometiendo la seguridad y el rendimiento de los cultivos. Aunque las plantas han desarrollado sistemas de tolerancia al arsénico esenciales para su supervivencia, su presencia en los suelos reduce el crecimiento de las mismas y conlleva graves pérdidas económicas en la agricultura. Esto es particularmente relevante en el cultivo de arroz, sobre todo en países asiáticos donde este cultivo es la fuente principal de alimento y la contaminación por arsénico es especialmente importante.
La publicación en la revista New Phytologyst del nuevo trabajo del grupo de Antonio Leyva en el CNB-CSIC identifica un factor de transcripción llamado GLABRA2 como un nuevo regulador de la tolerancia al arsénico en plantas. GLABRA2 es conocido por ser una proteína que regula la formación de tricomas (pelos en las hojas) y los pelos radiculares (en la raíz). Los pelos radiculares juegan un papel fundamental en la capacidad de absorción de nutrientes y arsénico. La identificación de GLABRA2 indica que este regulador es esencial para modular el número de pelos radiculares en función de la capacidad de detoxificación y expulsión de arsénico del interior de la planta.
El CSIC te llama esta noche: Noche Europea de los Investigadores/as 2024

Llevamos la mejor ciencia a la calle en un evento multitudinario que tendrá lugar en Espacio Fundación Telefónica. La Noche Europea de los Investigadores y las Investigadoras 2024 se celebrará el 27 de septiembre**, viernes, en el corazón de Madrid. Aquí tienes toda la información que necesitas.
** Las solicitudes de plazas para centros educativos (en horario de mañana) se abren el 16 de septiembre a las 9AM a través de este enlace: habrá 40 espacios para Primaria (5º y 5º) y 80 para Secundaria (entre 3ºESO y Bachillerato). El acceso a las actividades de la tarde es libre.
Resumen de enlaces para ampliar información:
- Oferta de actividades para Primaria
- Oferta de actividades para Secundaria
- Oferta de actividades en la feria científica para todos los públicos
- Oferta de talleres a puerta cerrada (con pases horarios)
- Programa de Monólogos científicos (pendiente)
Lanzan una guía para entender el envejecimiento celular
- Personal del CSIC participa en un artículo, publicado en la revista ‘Cell’, que compila la información disponible para identificar ‘in vivo’ las células envejecidas del organismo
La senescencia es un estado en el que las células envejecidas pierden la capacidad de dividirse y de contribuir al funcionamiento del organismo. Sin embargo, no desaparecen, sino que con el paso de los años se van acumulando en casi todos los órganos, donde segregan moléculas inflamatorias y enzimas que degradan los tejidos. Pese a que su descripción se produjo hace ya más de seis décadas, el proceso de senescencia celular fue siempre mal entendido por la dificultad para identificar correctamente las células que se encontraban en este estado. Ahora, un artículo publicado en la prestigiosa revista Cell presenta una guía que aporta pautas a los equipos de investigación para identificar las células senescentes in vivo.
Juan Carlos Acosta y Manuel Collado, investigadores del Consejo Superior de Investigaciones Científicas (CSIC), organismo dependiente del Ministerio de Ciencia, Innovación y Universidades (MICIU). “La senescencia celular es un proceso por el cual una célula dañada entra en un estado de reposo, sin dividirse, pero desencadenando señales que permitan a las células vecinas conocer el daño y promover la reparación del tejido”, explica Manuel Collado, investigador del Centro Nacional de Biotecnología en el CSIC (CNB-CSIC) y del Centro de Investigación en Medicina Molecular y Enfermedades Crónicas (CiMUS) de la Universidad de Santiago de Compostela. “Este mecanismo es muy eficaz a corto plazo, sin embargo, su acumulación a lo largo de la vida provoca la aparición de otras enfermedades crónicas asociadas con el envejecimiento”.
Announcing the 10th Edition of Science by Women Program

Women for Africa Foundation is excited to announce the launch of the 10th edition of the Science by Women program.
This initiative, known for its dedication to empowering African women in the scientific community, offers a unique opportunity for senior African women researchers to conduct their postdoctoral research in prestigious Spanish research centers, including the CNB.
Visualizan por primera vez cómo se produce la multiplicación del genoma del virus de la gripe
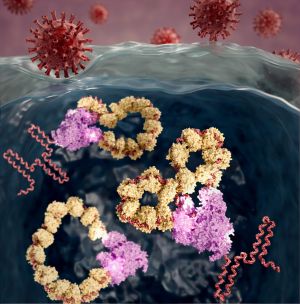
- Un equipo internacional liderado por el CSIC utiliza Microscopía de fuerza atómica de alta velocidad para ver a escala nanométrica cómo se mueven las moléculas virales para ejercer su acción
- Estos hallazgos mejoran la comprensión de los procesos fundamentales del ciclo de multiplicación del virus de la gripe A y pueden ayudar a crear medicamentos más eficaces
Un equipo internacional liderado por el Consejo Superior de Investigaciones Científicas (CSIC), organismo dependiente del Ministerio de Ciencia, Innovación y Universidades (MICIU), ha visualizado por primera vez cómo se produce la multiplicación del genoma del virus de la gripe. En el trabajo, publicado en la revista ACS Nano, se describe la visualización con técnicas avanzadas de imagen de la forma y los movimientos de las macromoléculas virales que se encargan de multiplicar el genoma viral. La colaboración entre equipos de investigación del CSIC, el instituto IMDEA Nanociencia y el Life Science Institute (Universidad de Kanazawa, Japón) ha permitido ver a escala nanométrica cómo se mueven las moléculas para ejercer su acción lo que puede ayudar a desarrollar tratamientos específicos.
El virus de la gripe A es una gran preocupación para la salud pública ya que es responsable de brotes de gripe estacionales y potenciales pandemias. En los últimos meses se ha descrito el aumento de la transmisibilidad entre mamíferos de la variante H5N1 del virus de la gripe aviar, cuyas mutaciones pueden hacer que nuevas cepas acaben afectando a humanos. En este escenario, comprender cómo este virus se replica y se propaga es crucial para desarrollar mejores tratamientos y medidas preventivas.
Jaime Martín-Benito, investigador del CSIC en el Centro Nacional de Biotecnología (CNB-CSIC) y uno de los responsables del estudio explica que “el virus de la gripe tiene un genoma formado por ocho moléculas de ARN (en vez de moléculas de ADN), y en su mecanismo de multiplicación participan complejos macromoleculares de ARN y proteínas (ribonucleoproteínas o RNPs) de distintos tamaños”.
“Para facilitar el estudio de su forma y movimiento, hemos empleado RNPs sintéticas generadas en el laboratorio con un tamaño concreto que alcanza para formar un anillo, de manera que todas son iguales. Con la Microscopía de fuerza atómica de alta velocidad hemos medido cómo se deforma el anillo mientras se hace la copia del genoma del virus”, detalla Martín-Benito.
Teresa Gil, Pablo Laborda y Carmen Campos, Premios Extraordinarios de Doctorado en la Universidad Autónoma de Madrid
Mercedes Ricote, XX Premio Ciencias de la Salud Fundación Caja Rural Granada por su trabajo sobre el papel de la leche materna en la maduración del corazón

- Esta investigación, publicada en Nature, describe el mecanismo por el que un ácido graso presente en la leche materna regula la maduración de las células cardiacas tras el nacimiento
- Los resultados podrían tener implicaciones terapéuticas en algunas patologías cardiovasculares
El artículo ‘El ácido g-linolénico en la leche materna regula la maduración metabólica cardiaca’, liderado por la doctora Mercedes Ricote y encabezado por la doctora Ana Paredes, se ha alzado con el XX Premio Ciencias de la Salud Fundación Caja Rural Granada dotado con 25.000 euros.
Identifican nuevos marcadores para el diagnóstico precoz de patologías hepáticas graves tras la curación de la hepatitis C
- Un estudio del CSIC describe genes como marcadores del estado del hígado tras la eliminación del virus de la hepatitis C crónica, que puede dar lugar a enfermedades hepáticas graves como la cirrosis o el carcinoma hepatocelular
- Aunque los tratamientos consiguen erradicar el virus en la práctica totalidad, se desconoce su incidencia en el desarrollo de carcinoma hepatocelular y este estudio podría facilitar el seguimiento de estos pacientes a largo plazo
Las nuevas combinaciones de fármacos antivirales frente al virus de la hepatitis C (VHC) han revolucionado la terapia contra la hepatitis crónica C, con la erradicación del virus en la práctica totalidad de los pacientes tratados. Aun así, se desconoce el impacto que la eliminación de la infección tiene sobre la incidencia de desarrollo de carcinoma hepatocelular (el tipo más común de cáncer de hígado) en estos pacientes, un proceso que no parece ser totalmente reversible a pesar de la curación de la infección por el VHC.
Un estudio liderado por personal investigador del Consejo Superior de Investigaciones Científicas (CSIC), organismo dependiente del Ministerio de Ciencia, Innovación y Universidades (MICIU), identifica un pequeño número de genes que se siguen expresando de forma anormal en células libres de la infección. Estas alteraciones son compatibles con las observadas en biopsias hepáticas de pacientes curados virológicamente y muestra el valor de los modelos de infección utilizados para la búsqueda de marcadores del estatus hepático post-curación en pacientes tras la eliminación de la infección, algo que podría facilitar el seguimiento de estos pacientes.
En el trabajo, dirigido por el investigador del CSIC en el Centro Nacional de Biotecnología (CNB-CSIC) Pablo Gastaminza, han utilizado dos modelos de infección persistente en cultivos celulares para analizar y comparar las diferencias a nivel de expresión génica tras la eliminación completa de la infección viral. Gastaminza destaca que, en el artículo publicado en Journal of Medical Virology, “describimos una reversión generalizada de la expresión de los genes relacionados con la infección después de la resolución de la misma, y como, sin embargo, un pequeño número de genes se siguen expresando de forma anormal en células que ahora están libres de la infección.”

