
Desvelada la estructura del adenovirus causante de un gran número de infecciones gastrointestinales infantiles
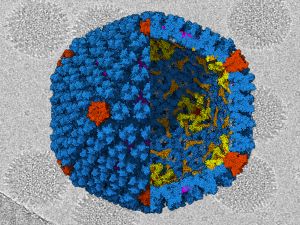
- Un equipo del CSIC revela características de la estructura del virus que podrían permitirle resistir e incluso aprovechar las condiciones ácidas del aparato digestivo para reproducirse
- Esta peculiaridad lo convierte en candidato para el desarrollo de vacunas u otros medicamentos estables en condiciones ácidas, o para administración por vía oral
Un equipo internacional, liderado por el Consejo Superior de Investigaciones Científicas (CSIC), ha revelado por primera vez la estructura del adenovirus de tipo 41 (Ad41), uno de los principales agentes virales que provocan la gastroenteritis infantil. El trabajo, publicado en Science Advances, sugiere que las diferencias observadas entre la envoltura proteica (cápsida) de este virus y la de otros adenovirus de tipo respiratorio podrían explicar la predilección del primero por el aparato digestivo. El conocimiento de la estructura del virus puede contribuir a desarrollar terapias dirigidas de manera exclusiva al tracto gastrointestinal y a nuevas vacunas basadas en los adenovirus, como las que se están desarrollando actualmente frente al virus SARS-CoV-2.
Descubren el mecanismo por el que las enterobacterias se adaptan a nuestro organismo
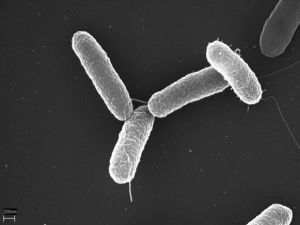
- Un grupo de investigación con participación del Instituto de Biomedicina de Valencia (IBV) y del Centro Nacional de Biotecnología de Madrid (CNB), ambos del CSIC, realiza la mejor caracterización del sistema que permite a la bacteria Salmonella sobrevivir en el cuerpo humano
- El hallazgo abre la puerta a diseñar nuevos fármacos antimicrobianos
Un grupo de investigación donde participan científicos del Instituto de Biomedicina de Valencia (IBV) y del Centro Nacional de Biotecnología (CNB), ambos del Consejo Superior de Investigaciones Científicas (CSIC), junto a la Universitat de València (Instituto Universitario Biotecmed) y la Universidad Autónoma de Madrid, han aclarado el mecanismo que emplea la bacteria patógena Salmonella para controlar la expresión de cientos de sus genes, entre ellos los que controlan la adaptación a nuestro organismo y, por tanto, su capacidad para infectarnos. El hallazgo, publicado en la revista Nucleic Acids Research, abre la puerta a diseñar nuevos fármacos antimicrobianos.
El trabajo describe con precisión el mecanismo de funcionamiento de la proteína RcsB de Salmonella para controlar la expresión de un número elevado de genes. RcsB es una proteína que se une al ADN para controlar la expresión de genes cuyos productos son necesarios para reorganizar la arquitectura de la envuelta celular en respuesta a daños externos. Esta proteína recibe señales de otras proteínas dispuestas en la envuelta y que actúan de antenas, formando todas ellas el denominado ‘sistema Rcs’, conservado en la familia de las enterobacterias (Enterobacteriaceae). Esta familia está formada por más de 100 géneros bacterianos que incluyen especies y serovares como Salmonella enterica serovar Typhi (S. typhi) o Shigella dysenteriae, causantes de la fiebre tifoidea y la disentería en humanos.
Nuevo método de obtención de imágenes celulares para comprender las infecciones virales
Identificados los genes que producen pelo en los frutos para adaptarse al clima

- La combinación de mutaciones en tres genes es necesaria para la aparición de pelo en los frutos de Arabidopsis, la planta más utilizada como modelo genético
- Esta combinación de genes ha evolucionado sólo en un linaje antiguo de la Península ibérica
- Este nuevo carácter podría ayudar a la adaptación de las plantas al cambio climático
Para identificar especies vegetales en Botánica, se utilizan claves de clasificación basadas en las características visuales que presentan las plantas. De esta forma se pueden distinguir especies cercanas que tiene una característica diferencial. En el caso de Arabidopsis thaliana, la planta más utilizada como modelo en investigación, una de sus características para identificarla es la presencia de pelos (tricomas) en las hojas pero no en los frutos.
Sin embargo, el nuevo trabajo del grupo dirigido por el investigador Carlos Alonso-Blanco en el Centro Nacional de Biotecnología del Consejo Superior de Investigaciones Científicas (CNB-CSIC) ha identificado poblaciones naturales de A. thaliana que desarrollan pelo en los frutos, lo que proporciona una nueva característica para esta planta. Su trabajo, publicado en la revista The Plant Cell detalla los mecanismos genéticos y moleculares que han permitido la aparición de pelo en los frutos de Arabidopsis.
Día internacional de la Mujer y la Niña 2021
El día 11 de febrero se celebra el Día Internacional de la Mujer y la Niña en la Ciencia. En el CNB-CSIC nos unimos a la Iniciativa 11 de Febrero, y este año, ademas del Seminario de Concha Monje, participamos en varias actividades coordinadas por los centros CSIC el campus de Cantoblanco:
- 9 febrero, 18:30. María José Rodríguez (IFT, USU). La increíble ciencia detrás de los agujeros negros, Gravedad y el premio Nobel de 2020
- 11 febrero, 14:00. Lluís Montoliu (CNB, CSIC). Dos investigadoras premiadas con el premio Nobel de Química de 2020 como promotoras de la revolución CRISPR.
- Escape Road: A la búsqueda de las científicas Nobel y no Nobel.
Casa de la Juventud de Tres Cantos. 13 febrero. Exposición y concurso. Acceso libre y gratuito.
Además:
- CNB: Webinar Concha Monje, Robótica: Retos y Aplicaciones, 11 Febrero 16:00
- #Youtubers por un día IFT
- ICMM: 8 febrero, 12:00. Esther Garcés, Silvia Donoso (ICM, CSIC). Plan de Igualdad de Género como instrumento para el avance en la igualdad en la investigación, el ejemplo en el Institut de Ciències del Mar
- ICMM-UAM: Talleres en la Facultad de Profesorado de la UAM. 17 febrero, 12:00 y 15:30. Elena Bascones (ICMM, CSIC). Por qué las niñas no estudian ciencias técnicas y qué podemos hacer en clase.
- ICMAT: 11 febrero, 12:00. Marta Folgueira López (UCM). Constelaciones matemáticas.
- ICV. Ciclo de webinarios, ICV en Femenino, 8 de Febrero y 12 de marzo
- CIAL. Quiz Online, Webinars, talleres
- Escape Road: A la búsqueda de las científicas Nobel y no Nobel. Campus UAM

Identificada una nueva estrategia para frenar las resistencias a antibióticos
- Un trabajo del CNB-CSIC y el IRyCIS se centra en los elementos genéticos móviles de las bacterias (plásmidos) como herramientas para frenar la expansión de las resistencias
- Los plásmidos son el principal mecanismo de adquisición de resistencia en patógenos bacterianos de relevancia clínica
- Al ritmo actual, las infecciones causadas por bacterias resistentes a los antibióticos disponibles pueden convertirse en la mayor causa de mortalidad para el año 2050
La resistencia de las bacterias a los antibióticos es uno de los principales problemas actuales de salud pública. De acuerdo con estimaciones recientes, aproximadamente 700.000 personas mueren cada año en el mundo como consecuencia de infecciones resistentes a los antibióticos y, de no revertirse la tendencia actual de diseminación de la resistencias, en el 2050 las infecciones resistentes podrían convertirse en la primera causa de mortalidad en el mundo. Por este motivo, existe una necesidad urgente de desarrollar nuevas estrategias terapéuticas destinadas a contrarrestar las bacterias resistentes a antibióticos.
Investigadores del Centro Nacional de Biotecnología perteneciente al Consejo Superior de Investigaciones Científicas (CNB-CSIC), en colaboración con el Instituto Ramón y Cajal de Investigación Sanitaria (IRYCIS) del Hospital Universitario Ramón y Cajal en Madrid, han descrito una nueva estrategia basada en explotar la respuesta fisiológica que inducen los plásmidos en las bacterias y lograr así revertir la evolución de las resistencias a los antibióticos. Los detalles del trabajo, publicado en la revista eLife, podrían sentar las bases para el desarrollo de nuevas terapias contra las bacterias portadoras de estos plásmidos de resistencia, basándose en un fenómeno de la sensibilidad colateral asociada a ellos.
El CSIC y la empresa Ercros diseñarán un proceso de producción de polímeros de base biológica

- La plataforma SusPlast del CSIC y la compañía química Ercros acuerdan un proyecto de I+D para implementar una metodología de producción de estos materiales
- Estos polímeros se caracterizan por su alta susceptibilidad a la biodegradación y son útiles en varios sectores industriales para aplicaciones de corta y larga duración
- Lograr un bioproceso de producción es clave para disponer de materiales sostenibles de origen biológico renovable
El Consejo Superior de Investigaciones Científicas (CSIC) y la empresa química Ercros han firmado un acuerdo para diseñar un proceso de producción de biopolímeros bacterianos. Estos materiales son útiles tanto para usos de corta como de larga duración y se aplican en sectores industriales tan diversos como el del envase y embalaje, el agrícola y el cosmético.
“Conseguir polímeros de base biológica renovable es importante para disponer de materiales sostenibles que permitan avanzar hacia la economía circular. La nueva tecnología abaratará los costes de producción de estos biopolímeros al disponer de un proceso más eficiente, más sostenible (ya que emplea materias primas de baja huella de carbono) y que aporta ventajas derivadas de las prestaciones de los materiales”.
En este proyecto participan dos equipos de investigación del CSIC agrupados en la plataforma temática interdisciplinar SusPlast (Interdisciplinary Platform for Sustainable Plastics towards a Circular Economy). Los dos equipos son el Grupo de Biotecnología de Polímeros, del Centro de Investigaciones Biológicas Margarita Salas (CIB-CSIC), dirigido por Auxiliadora Prieto, que también coordina la plataforma; y el Grupo de Biotecnología de Sistemas, del Centro Nacional de Biotecnología (CNB-CSIC), dirigido por Juan Nogales.
La vacuna del CSIC más adelantada para la covid-19 muestra una eficacia del 100% en ratones

- El proyecto de Mariano Esteban y Juan García Arriaza, del CNB-CSIC, que utiliza una variante del virus que sirvió para erradicar la viruela, se publica en ‘Journal of Virology’
- La producción de la vacuna se está desarrollando en colaboración con la biotecnológica española Biofabri, del grupo Zendal, para comenzar los ensayos clínicos
El candidato vacunal MVA-CoV-2-S, que usa como vehículo el virus vaccinia modificado de Ankara (MVA) para transportar una proteína del SARS-CoV-2 (la proteína S) que estimule la defensa inmunitaria contra el coronavirus, ha probado su eficacia en modelos animales, y está por tanto listo para seguir avanzando hacia las pruebas clínicas, según se publica en la prestigiosa revista Journal of Virology.
El proyecto de vacuna para el SARS-CoV-2 que dirigen los virólogos Mariano Esteban y Juan García Arriaza es el más adelantado de las tres vacunas contra la covid-19 que se están desarrollando en el Consejo Superior de Investigaciones Científicas (CSIC).
Entrevista a Mario Mellado, director del CNB
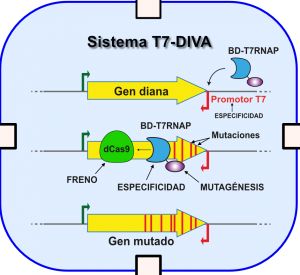
Diseñan un nuevo sistema de mutagénesis in vivo para acelerar la evolución de proteínas con interés biotecnológico
- Las técnicas que permiten acelerar la evolución de proteínas son herramientas muy útiles en procesos de biomedicina y biotecnología
- El nuevo sistema T7-DIVA, diseñado por investigadores del CNB-CSIC, permite el rápido desarrollo de variedades sintéticas de anticuerpos y proteínas relacionadas con la resistencia a antibióticos entre otras aplicaciones
Investigadores del Centro Nacional de Biotecnología del Consejo Superior de Investigaciones Científicas (CNB-CSIC) desarrollan un nuevo sistema de mutagénesis in vivo que permite generar variantes de proteínas de una forma rápida y sencilla en bacterias, y se puede adaptar para su utilización en levaduras y otras células eucariotas. El nuevo sistema, publicado en Nature Communications, permite la selección de variantes de una forma continua y con poca manipulación.
Las técnicas de evolución molecular dirigida en el laboratorio permiten generar variantes de proteínas con interés biotecnológico o biomédico como anticuerpos o enzimas terapéuticas con funciones mejoradas o incluso nuevas de manera mucho más eficaz. Hasta hace poco tiempo, la mayoría de las técnicas de evolución dirigida se realizaban in vitro, en procesos lentos y tediosos, por lo que en los últimos años hay un interés creciente por el desarrollo de técnicas de evolución molecular in vivo.
Ahora, la nueva técnica desarrollada por el grupo del investigador Luis Ángel Fernández en el Centro Nacional de Biotecnología del CSIC permite dirigir las mutaciones de manera específica a la región génica de interés dentro de las células, expresar las variantes y seleccionarlas de una forma rápida, continua y sin mucha manipulación.


