
Descrito cómo el virus del resfriado libera su información genética
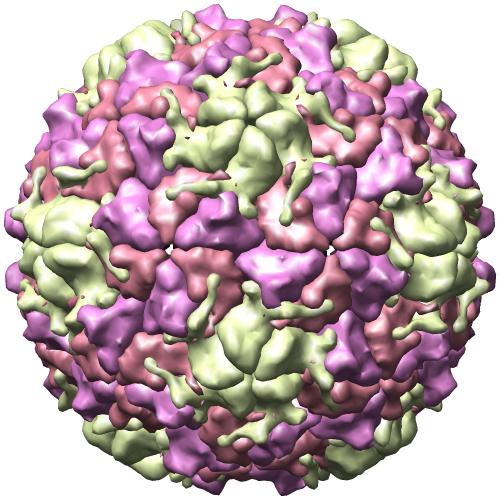 La profesora de investigación del CSIC Núria Verdaguer lleva años estudiando en Barcelona este proceso mediante cristalografía de rayos X. En su trabajo más reciente ha establecido la estructura atómica de la cápside vacía del Rhinovirus humano tipo 2.
La profesora de investigación del CSIC Núria Verdaguer lleva años estudiando en Barcelona este proceso mediante cristalografía de rayos X. En su trabajo más reciente ha establecido la estructura atómica de la cápside vacía del Rhinovirus humano tipo 2.En colaboración con el experto de biología estructural de virus del Centro Nacional de Biotecnología del CSIC José Ruiz Castón y Dieter Blass del Vienna Biocenter, acaban de publicar un trabajo en el que muestran los cambios conformacionales de la cáspide mediados por el pH ácido. Estos cambios resultan en la formación de canales en la pared de la cápside que facilitarían la salida del ARN viral.
Las reorganizaciones estructurales de las proteínas VP1, VP2 y VP3 de la cápside pueden apreciarse en el vídeo. Con la nueva organización de las proteínas de la cápside aparecen unos poros por donde, como explica Castón, "saldrá el material genético infeccioso al citoplasma de la célula". Un paso imprescindible para que se multiplique el virus.
La hipermutagenicidad de Pseudomonas aeruginosa
 En su laboratorio del Centro Nacional de Biotecnología, Alexandro Rodríguez-Rojas y Jesús Blázquez, en colaboración con Antonio Oliver del Hospital Son Espases, han analizado las causa por las que cuando la bacteria Pseudomonas aeruginosa infecta a los enfermos que se encuentran en los hospitales se genera "un alto número de mutantes en la población, incluyendo individuos con resistencia a la mayoría de los antibióticos". De este modo, una vez establecidas en el pulmón de pacientes con enfermedades crónicas, las bacterias con mutaciones son
seleccionadas por los antibióticos con los que se están tratando,
originando, como explican los autores, "infecciones crónicas casi imposibles
de erradicar".
En su laboratorio del Centro Nacional de Biotecnología, Alexandro Rodríguez-Rojas y Jesús Blázquez, en colaboración con Antonio Oliver del Hospital Son Espases, han analizado las causa por las que cuando la bacteria Pseudomonas aeruginosa infecta a los enfermos que se encuentran en los hospitales se genera "un alto número de mutantes en la población, incluyendo individuos con resistencia a la mayoría de los antibióticos". De este modo, una vez establecidas en el pulmón de pacientes con enfermedades crónicas, las bacterias con mutaciones son
seleccionadas por los antibióticos con los que se están tratando,
originando, como explican los autores, "infecciones crónicas casi imposibles
de erradicar".Estas variantes presentan diferentes mutaciones que les permiten adaptarse de una forma u otra tanto a la acción de los antibióticos como a las defensas del enfermo. Entender el origen de estas mutaciones es importante para poder desarrollar estrategias que permitan luchar contra las infecciones crónicas.
Una de las causas de la enorme cantidad de mutaciones son las especies reactivas de oxígeno generadas por los propios glóbulos blancos para luchar contra la infección. Otra son las cepas de esta bacteria que suelen encontrarse en los hospitales. Por lo general carecen de una serie de proteínas que en condiciones normales evitarían la generación de mutaciones.
Con estos datos, la solución sería el tratamiento con una combinación de antibióticos. Combinaciones que ya se hacen y que como explica en su última publicación este grupo del CNB, no son muy efectivas. Y aunque esto indica que hay que seguir investigando para saber con mayor exactitud qué está pasando con este tipo de mutaciones, abre la puerta a investigar nuevas estrategias, como la reducción de la capacidad de mutación de estas bacterias.
Reunión científica sobre el virus de la Hepatitis C
 Gracias al patrocinio de la Sociedad Española de Virología (SEV) y del propio CSIC, la reunión podrá contar con la presencia de
Profesor Charles Rice, Director del Centro de Estudio de la Hepatitis C de la Rockefeller University en Nueva York.
Gracias al patrocinio de la Sociedad Española de Virología (SEV) y del propio CSIC, la reunión podrá contar con la presencia de
Profesor Charles Rice, Director del Centro de Estudio de la Hepatitis C de la Rockefeller University en Nueva York.La reunión constará de tres sesiones que cubrirán las áreas BÁSICA, CLÍNICA y DIAGNÓSTICO–EPIDEMIOLÓGICA en el que habrá lugar para la participación de los ponentes cuyo resumen sea seleccionado para la presentación. Cada uno de éstos bloques será presentado por los Coordinadores de Área del Grupo de Hepatitis Víricas de la SEV que nos darán su visión general sobre el estado actual y sobre los desafíos más relevantes en cada una de las áreas.
Además se contará con la presencia de un panel de expertos que participarán en una mesa redonda en la que se presentará el Grupo de Hepatitis Víricas de la Sociedad Española de Virología y en la que se discutirán diversos aspectos del estudio de estos virus.
La inscripción al Workshop es gratuita pero obligatoria. Las inscripciones se pueden hacer por correo electrónico en la dirección de correo electrónico del Dr. Gastaminza, indicando el nombre, correo electrónico, afiliación y si se desea presentar una breve comunicación oral. Se ruega nos comunique su participación antes del 15 de marzo para poder estimar el número de asistentes con suficiente antelación. Si desea exponer brevemente su trabajo (10 minutos, más 3 minutos de preguntas), por favor envíe (antes del 5 de marzo) un título, un resumen de 250 palabras así como la lista de autores y su afiliación junto con el correo de registro. Se ruega que tanto los resúmenes como las comunicaciones orales sean en INGLÉS. Debido a restricciones de tiempo, solo algunos resúmenes podrán ser presentados. Los autores que presenten su trabajo tendrán un año de suscripción gratuita a la SEV si aún no son socios.

Detectan en tiempo real la actividad de una proteína utilizando micropalancas
La colaboración entre el grupo de Javier Tamayo, del Instituto de Microelectrónica de Madrid, con el de José L. Carrascosa en el Centro Nacional de Biotecnología del CSIC ha permitido la detección en tiempo real del movimiento de una proteína encargada de empaquetar el ADN viral dentro de la cápsida.
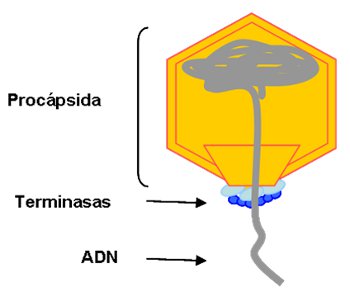 Como explica el Dr. Carrascosa, para que un virus pueda infectar es totalmente imprescindible que empaquete su ADN dentro de la cabeza viral. Para que se pueda llevar a cabo el empaquetamiento, los virus cuentan con una especie de motor, la terminasa, que además de consumir energía sufre cambios de conformación, es decir, se mueve.
Como explica el Dr. Carrascosa, para que un virus pueda infectar es totalmente imprescindible que empaquete su ADN dentro de la cabeza viral. Para que se pueda llevar a cabo el empaquetamiento, los virus cuentan con una especie de motor, la terminasa, que además de consumir energía sufre cambios de conformación, es decir, se mueve.
Empleando una técnica biofísica denominada micropalancas, los científicos del CSIC han sido capaces de detectar en tiempo real el cambio que sufre este motor molecular. Con apenas medio milímetro de largo, las micropalancas cuentan con un láser a su lado que puede registrar los movimientos provocados por las terminasas.
Al añadirle ATP (la "gasolina" de este motor molecular), las terminasas que están adheridas a la micropalanca se mueven. Y esto es lo que han sido capaces de detectar a tiempo real, un movimiento escalonado de la micropalanca que se corresponde con la actividad de multitud de motores sobre su superficie. Como recalca la investigadora predoctoral María I. Daudén, gracias a estas micropalancas han sido capaces de "detectar el cambio conformacional provocado por la hidrólisis de ATP".

El Dr. Johann Mertens nos explica que los resultados, publicados en la revista Nanotechnology, validan la capacidad de nuestro sistema híbrido (bio-mecánico) para transformar energía química en movimiento. Además, añade que ha supuesto una mejora en sensibilidad en la detección de la actividad de estas proteínas de dos órdenes de magnitud respecto a los métodos bioquímicos tradicionales.
Estos resultados son un paso importante en la aplicación de las micropalancas para el desarrollo de nuevas nanomáquinas y sensores, que están siendo objeto de creciente interés en diferentes campos de la nanotecnología.
XIX Jornadas de Navidad del CNB
Estudiantes La Caixa
El estudiante de doctorado indio Aneesh Vijayan lleva desde XXX de 2008 trabajando en el laboratorio de Mariano Esteban con la intención de aumentar la efectividad de las vacunas actuales contra la malaria mediante el uso de proteínas que mejoren la respusta inmunitaria. Sus trabajos han demostrado en ratones que la proteína que han generado es capaz de aumentar la eficacia de la vacuna contra la malaria.
5ª convocatoria del Programa Internacional de becas de doctorado La Caixa / CNB
Por quinto año consecutivo, la Fundación "la Caixa" convoca 10 becas de cuatro años para desarrollar un doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC.
 Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas de becas para cursar estudios de postgrado tanto en España como en el extranjero.
Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas de becas para cursar estudios de postgrado tanto en España como en el extranjero.
Por quinto año consecutivo, la Fundación "la Caixa" convoca 10 becas de cuatro años para desarrollar un doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC, el mayor centro español y uno de los centros líderes en el mundo en el área de Biotecnología, Biología Molecular y Biomedicina.
De entre los más de 500 solicitantes, el Comité de Selección entrevistó a treinta candidatos y ha elaborado la siguiente lista para conceder las 10 becas:
|
Suplentes:
|
|
5º Ciclo de Seminarios Junior
El CNB recibe financiación de los Grand Challenges Explorations
La Fundación Bill & Melinda Gates acaba de anunciar que financiará al Centro Nacional de Biotecnología del CSIC (CNB) a través de sus Grand Challenges Explorations. Este programa, permite ensayar ideas novedosas y originales que intentan resolver problemas de salud global. En el caso concreto del CNB, el investigador Mark van Raaij intentará desarrollar un método para generar virus capaces de atacar a las bacterias intestinales patógenas.
 Los fondos de las Grand Challenges Explorations están destinados a investigadores de todo el mundo para que puedan explorar ideas poco ortodoxas que aborden problemas globales de salud. Entre los 110 proyectos financiados en esta convocatoria se encuentra el de van Raaij.
Los fondos de las Grand Challenges Explorations están destinados a investigadores de todo el mundo para que puedan explorar ideas poco ortodoxas que aborden problemas globales de salud. Entre los 110 proyectos financiados en esta convocatoria se encuentra el de van Raaij.
La Fundación Bill & Melinda Gates “cree en el poder de la innovación”, comenta Chris Wilson, director de Global Health Discovery de esta fundación. “Con tan solo un proyecto valiente se podría comenzar a solucionar los mayores problemas de salud y desarrollo. Las Grand Challenges Explorations pretenden encontrar esas ideas originales y ayudar a los científicos y a los emprendedores a llevar a cabo ese tipo de proyectos novedosos que pueden ser esenciales para acabar con la polio, el sida o mejorar las condiciones sanitarias de la población”. Los proyectos que están recibiendo su financiación resultan prometedores a la hora de afrontar retos globales de salud para los que todavía no existe una solución. Los hay desde los que tratan de encontrar métodos para eliminar o controlar enfermedades infecciosas como la polio o el sida hasta aquellos que intentan desarrollar nuevas tecnologías para mejorar las condiciones sanitarias de la población.
Por la gran frecuencia con la que las bacterias desarrollan resistencias frente a los antibióticos, los científicos buscan alternativas como puedan ser las vacunas o el uso de proteínas bacteriolíticas. Además, aprovechando la existencia natural de virus que sobreviven en el intestino e infectan a las bacterias, los llamados bacteriófagos son otra opción a la hora de luchar contra las bacterias intestinales. En los tratamientos actuales con bacteriófagos que se han ensayado se emplean combinaciones de uno o varios de estos enemigos naturales de las bacterias. Lo que propone hacer van Raaij es generar mutantes de los bacteriófagos T4 y T7 para que, de una forma específica, reconozcan, infecten y maten a las enterobacterias.
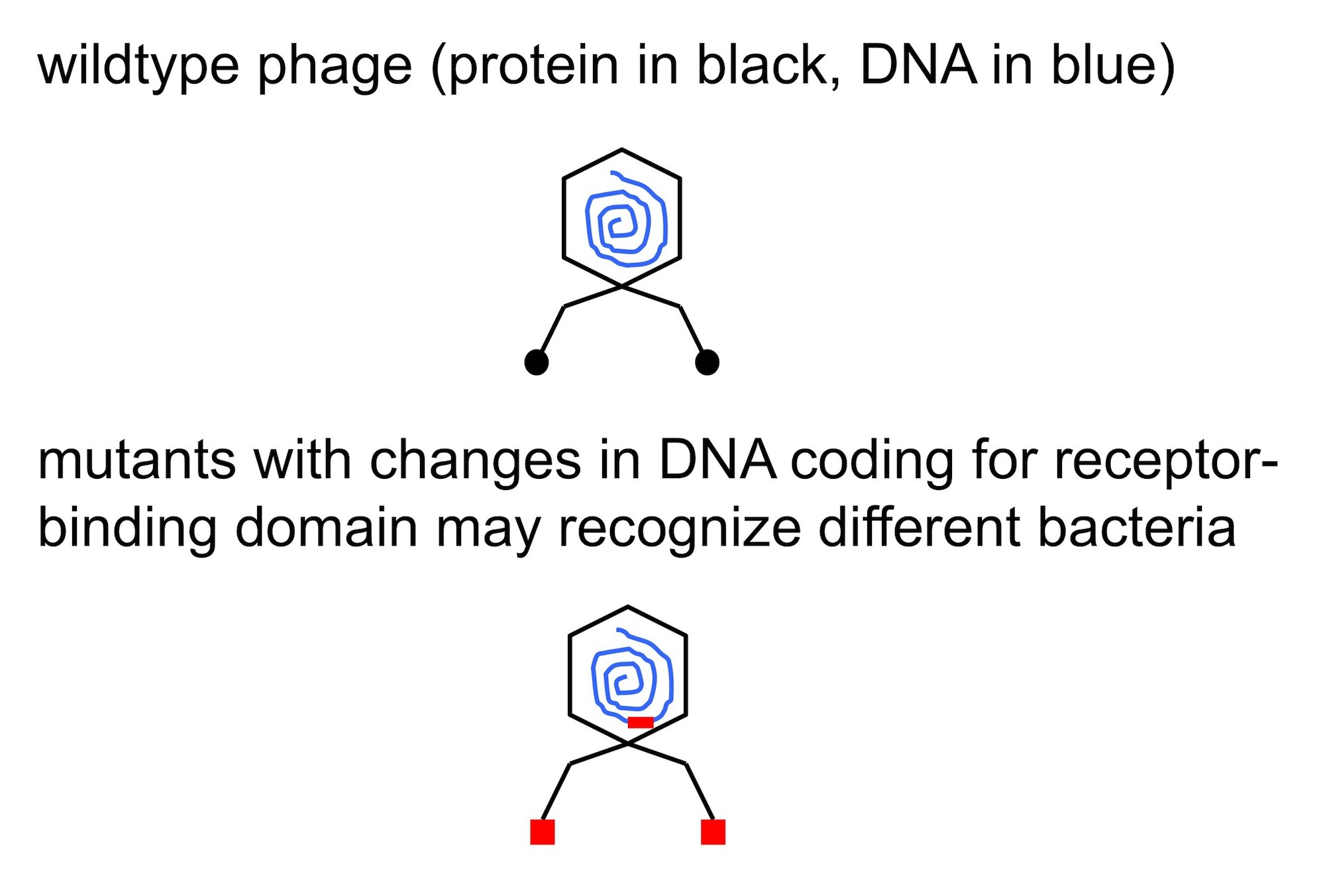 Los bacteriófagos T4 y T7, que en condiciones naturales afectan sólo a la bacteria Escherichia coli, son desde hace décadas un conocido sistema modelo de los biólogos moleculares. Sin embargo, hasta que van Raaij no publicó sus estudios en 2010 y 2011, no se conocía con precisión la estructura de las fibras mediante las que se unen a las bacterias. Ahora, en su laboratorio del CNB pretende generar bacteriófagos que contengan mutaciones aleatorias en las zonas que determinan su unión a las bacterias. Con los miles de mutantes que planean obtener, tendrán que ir analizando la especificidad con la que se unen a las diferentes bacterias. Una vez que hayan detectado los mutantes que eliminan específicamente a las bacterias patógenas intestinales, los producirán en grandes cantidades para poder ensayar su uso como posible tratamiento.
Los bacteriófagos T4 y T7, que en condiciones naturales afectan sólo a la bacteria Escherichia coli, son desde hace décadas un conocido sistema modelo de los biólogos moleculares. Sin embargo, hasta que van Raaij no publicó sus estudios en 2010 y 2011, no se conocía con precisión la estructura de las fibras mediante las que se unen a las bacterias. Ahora, en su laboratorio del CNB pretende generar bacteriófagos que contengan mutaciones aleatorias en las zonas que determinan su unión a las bacterias. Con los miles de mutantes que planean obtener, tendrán que ir analizando la especificidad con la que se unen a las diferentes bacterias. Una vez que hayan detectado los mutantes que eliminan específicamente a las bacterias patógenas intestinales, los producirán en grandes cantidades para poder ensayar su uso como posible tratamiento.
Acerca de las Grand Challenges Explorations
Las Grand Challenges Explorations es una iniciativa de la Fundación Bill & Melinda Gates dotada con 100 millones de dólares. En funcionamiento desde 2008, las Grand Challenge Explorations han financiado ya a casi 500 científicos de 40 países diferentes. El programa está abierto a la participación de investigadores de cualquier disciplina y organización. La selección se lleva a cabo dos veces al año mediante un proceso ágil y rápido de evaluación y concesión de los proyectos en los que no se necesitan resultados experimentales previos, y tan solo hay que rellenar online una solicitud de dos páginas. Inicialmente, los proyectos elegidos cuentan con una financiación de 100.000 dólares que, en el caso de tener éxito, podrían recibir posteriormente hasta 1 millón de dólares.

