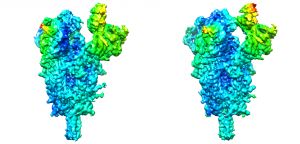
José María Carazo, investigador del Centro Nacional de Biotecnología y uno de los responsables del trabajo, destaca que “mediante los nuevos desarrollos propuestos en análisis de imagen hemos podido comenzar a entender la flexibilidad de la espícula infectiva del virus, detectando algunas de las bisagras moleculares que facilitan su entrada en la célula. Para lograrlo hemos estudiado cientos de miles de imágenes de microscopía electrónica en condiciones criogénicas, unido grupos de trabajo en España y Estados Unidos, utilizado recursos computaciones en diferentes instituciones, logrando resultados impensables sólo hace unos años y en un tiempo record” remarca Carazo. “De esta forma, hemos podido identificar el movimiento que sigue la proteína S para fusionarse con las membranas celulares”.
Roberto Melero, primer autor del trabajo, junto con Carlos Oscar Sanchez Serrano, señala, “este trabajo prueba la existencia de una flexibilidad continua y característica en la proteína S del SARS-CoV2 que no habíamos sido capaces de detectar previamente, y que podría ser útil en el diseño de nuevas terapias dirigidas a bloquear la entrada del virus en la célula”.
En la obtención de estos resultados han colaborado el Centro Nacional de Biotecnología y Centro de Química Física Rocasolano pertenecientes ambos al Consejo Superior de Investigaciones Científicas (CNB-CSIC y IFQR-CSIC) en colaboración con la Universidad Autónoma de Madrid, la Universidad de Yale y la Universidad de Texas en Estados Unidos, y han contado con el apoyo del Instituto de Física de Cantabria, del Centro de Supercomputación de Barcelona así como de la Infraestructura Europea de Biología Estructural “Instruct-ERIC”.
Más información:
Nota de prensa del CSIC Comunicación
Artículo científico
Continuous flexibility analysis of SARS-CoV-2 spike prefusion structures. R Melero, COS Sorzano, B Foster, JL Vilas, M Martínez, R Marabini, E Ramírez-Aportela, R Sánchez-García, D Herreros, L del Caño, P Losana, YC Fonseca-Reyna, P Conesa, D Wrapp, P Chacón, JS McLellan, HD Tagareb, JM Carazo. IUCrJ Volume 7| Part 6| November 2020 DOI: https://doi.org/10.1107/S2052252520012725