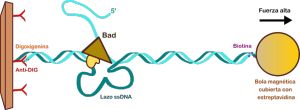
Carolina Carrasco, investigadora del CNB-CSIC, destaca la utilidad de técnicas de análisis de molécula única como las pinzas magnéticas “para estudiar cómo Bad recorre el ADN, las pausas y retrocesos que realiza así como la velocidad a la que lo hace”. “Uno de los descubrimientos más llamativos es que Bad es una enzima tremendamente rápida capaz de moverse por el ADNa una velocidad de 200 pares de bases por segundoa lo largo de 4000 pares de bases a temperatura ambiente sin perder su unión al ADN que desenrolla”. Clara Aicart, colaboradora en el trabajo remarca que durante el proceso, Bad se engancha a la hebra complementaria del ADN, de modo que se va formando un lazo de ADN de cadena sencilla, una característica emergente de algunas helicasas procesivas, que podría servir para ayudar a mantener la hélice desenrollada y facilitar procesos de reparación por recombinación homóloga.
La enzima Bad bacteriana supondrá un excelente sistema modelo para estudiar y entender las propiedades bioquímicas de otras enzimas similares a DNA2 así como de proteínas motoras cuya actividad conlleva la formación de lazos en el ADN empleando técnicas de molécula única.
Respecto a su función fisiológica, no parece probable que su papel sea exactamente el mismo al de DNA2. Una de las hipótesis que se baraja es que Bad sea una subunidad de una nueva clase de enzimas de restricción que podrían desenrollar y degradar el ADN de manera simultánea.
Más información:
Bulk and single molecule analysis of a bacterial DNA2-like helicase nuclease reveals a single-stranded DNA looping motor. Wilkinson OJ, Carrasco C, Aicart-Ramos C, Moreno-Herrero F, Dillingham MS. Nucleic Acid Res 2020 DOI: 10.1093/nar/gkaa562 10.1093/nar/gkaa562