Los plásmidos son moléculas de ADN circulares presentes en bacterias que se replican (duplican) de forma independiente del cromosoma bacteriano. La transmisión de plásmidos entre bacterias puede suponer la ganancia o la perdida de una función determinada en la célula receptora. Por ejemplo, algunos genes contenidos en plásmidos son capaces de conferir resistencia a antibióticos, y la transmisión de estos genes puede ser beneficiosa para la supervivencia bacteriana, ya que se convertirán en resistentes a esos antibióticos.
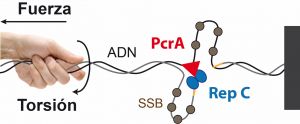
El sistema más habitual de duplicación de estos plásmidos se llama replicación por circulo rodante (RCR). Este proceso requiere la unión de la proteína Rep a una secuencia determinada del ADN, el corte de una de las cadenas del ADN y la separación de la doble cadena a medida que la helicasa PcrA se mueve por una de ellas. Sin embargo, el mecanismo por el cual se inicia la replicación y se desenrolla la doble hélice del ADN no se conoce con detalle.
Investigadores del Centro Nacional de Biotecnología, en colaboración con un grupo de científicos de la Universidad de Pittsburgh (Estados Unidos) han sido capaces de observar a nivel molecular la interacción de las proteínas Rep y PcrA en el proceso de replicación de plásmidos portadores de genes de resistencia a antibióticos.
El trabajo, publicado en la revista Nucleic Acids Research, revela que el proceso de la replicación se inicia con el corte de la doble hebra del ADN por RepC, y que ésta proteína recluta a la helicasa PcrA formando un complejo. Utilizando técnicas de microscopía que permiten analizar el proceso a nivel de molécula única, han observado cómo estas proteínas juntas son capaces de desenrollar el ADN a lo largo de miles de pares de bases sin soltarse de la molécula de ADN. Además, este motor molecular se mueve a velocidades de hasta 50 pares de bases por segundo, pero no lo hace de forma continua, sino que realiza pausas de forma aleatoria.
Fernando Moreno-Herrero, director del trabajo explica que “este trabajo demuestra el equilibrio entre fuerzas y torsiones que sufre una molécula de ADN para que se inicie la replicación y cómo éstas afectan a la unión de la proteína Rep y a su socio, el motor molecular PcrA, que desenreda el ADN para que otras máquinas proteínas lo copien a continuación”.
Más información:
Nucleic Acids Research, gkz1200, Dynamics of DNA nicking and unwinding by the RepC–PcrA complex.Carolina Carrasco, Cesar L Pastrana, Clara Aicart-Ramos, Sanford H Leuba, Saleem A Khan, Fernando Moreno-Herrero
Published: 13 January 2020