- El trabajo se basa en la comprensión de la evolución para diseñar tratamientos frente a bacterias como Pseudomonas aeruginosa, con baja sensibilidad a los antibióticos
- Las infecciones provocadas por bacterias resistentes a múltiples fármacos constituyen una preocupación creciente para la salud humana
Los antibióticos han supuesto uno de los mayores avances para la medicina moderna. De ahí que la resistencia a estos medicamentos sea actualmente una de las principales amenazas para la salud mundial, según la Organización Mundial de la Salud (OMS). Este problema es consecuencia de la capacidad de adaptación y evolución de las bacterias patógenas cuando se enfrentan a los antibióticos.
Plant Cell Physiol. 2020 Mar 27. pii: pcaa041
Fernández-Santos R, Izquierdo Y, López A, Muñiz L, Martínez M, Cascón T, Hamberg M, Castresana C.
Abstract
Lipid Droplets (LDs) have classically been viewed as seed storage particles, yet they are now emerging as dynamic organelles associated with developmental and stress responses. Nevertheless, their involvement in plant immunity has still been little studied. Here, we found LD accumulation in Arabidopsis thaliana leaves that induced a hypersensitive defense response (HR) after Pseudomonas infection. We established a protocol to reproducibly isolate LDs and to analyze their protein content. The expression of GFP fusion proteins in Nicotiana benthamiana and in transgenic Arabidopsis lines validated the LD localization of glycerol-3-phosphate-acyltransferase 4 (GPAT4) and 8 (GPAT8), required for cutin biosynthesis. Similarly, we showed LD localization of α-dioxygenase1 (α-DOX1) and caleosin3 (CLO3), involved in the synthesis of fatty acid derivatives, and that of phytoalexin deficient 3 (PAD3), which is involved in camalexin synthesis. We found evidence suggesting the existence of different populations of LDs, with varying protein content and distributions. GPAT4 and GPAT8 were associated with LDs inside stomata and surrounding cells of untreated leaves, yet they were mainly confined to LDs in guard cells after bacterial inoculation. By contrast, α-DOX1 and PAD3 were associated with LDs in the epidermal cells of HR-responding leaves, with PAD3 mostly restricted to cells near dead tissue, while CLO3 had a more ubiquitous distribution. As such, the nature of the proteins identified, together with the phenotypic examination of selected mutants, suggest that LDs participate in lipid changes and in the production and transport of defense components affecting the interaction of plants with invading pathogens.
doi: 10.1093/pcp/pcaa041
Los microorganismos resistentes a antibióticos necesitan readaptar sus procesos metabólicos para no perder capacidad de supervivencia. Bloquear estos procesos de reorganización de la maquinaria interna de las bacterias puede ofrecer nuevas estrategias en la lucha contra los patógenos resistentes a antibióticos.
Las bacterias que atacan a las plantas, además de secretar toxinas, inyectan en las células vegetales proteínas efectoras que favorecen la infección. Algunas cepas de Pseudomonas producen una toxina llamada coronatina que desorganiza la fisiología de la célula e impide el funcionamiento de sus defensas. La coronatina imita el funcionamiento de una de las hormonas encargadas de activar las defensas contra los hongos, el jasmonato. Pero, al mismo tiempo, promueve el crecimiento bacteriano al inhibir los mecanismos de defensa dependientes del ácido salicílico, los cuales son fundamentales para resitir las infecciones por Pseudomonas.
El grupo dirigido por Roberto Solano en el Centro Nacional de Biotecnología del CSIC intenta elucidar cómo las cepas de Pseudomonas que no producen coronatina, la mayoría de ellas, son capaces de alterar las hormonas de las plantas y qué mecanismos han evolucionado para que puedan infectarlas.
Aparte de las fitotoxinas, las cepas de Pseudomonas producen hasta 30 proteínas efectoras de tipo III distintas que son capaces de suprimir de forma activa las defensas de las plantas infectadas. Para el caso concreto de uno de estos efectores, HopX1, Solano acaba de encontar en su laboratorio del CNB que en la planta modelo Arabidopsis, tanto la expresión exógena de HopX1 como la infección natural por cepas de Pseudomonas que producen este efector, promueven el crecimeinto bacteriano de manera similar a la coronatina: activando la expresión de los genes dependientes del jasmonato. Sus resultados indican que HopX1 es una cisteína proteasa que imita los efectos de la coronatina al degradar a represores esenciales en el funcionamiento de la vía de señalización del jasmonato, las llamadas proteínas jasmonate-ZIM domain (JAZ).
Estos resultados, publicados en la revista PLoS Biology, muestran un mecanismo hasta ahora desconocido por el que los patógenos manipulan los puntos clave de la respuesta hormonal que usan las plantas para protegerse frente a las infecciones.
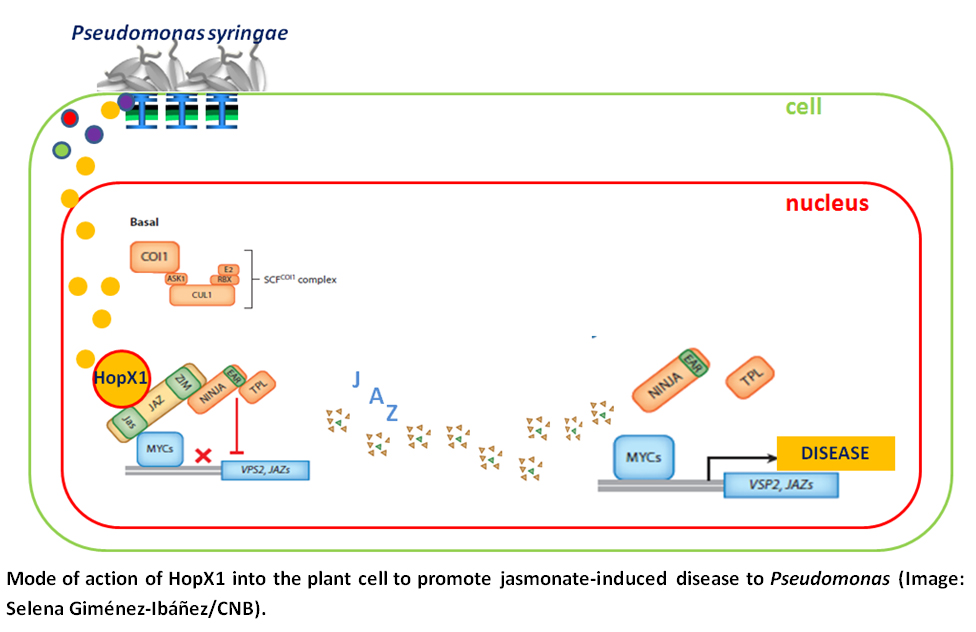
- Gimenez-Ibanez S, Boter M, Fernández-Barbero G, Chini A, Rathjen JP, Solano R. The bacterial effector HopX1 targets JAZ transcriptional repressors to activate jasmonate signaling and promote infection in Arabidopsis. PLoS Biol. 2014; 12(2): e1001792.
BMC Syst Biol. 2011 Nov 11;5:191
Silva-Rocha R, de Jong H, Tamames J, de Lorenzo V.
 The genetic network of the TOL plasmid pWW0 of the soil bacterium Pseudomonas putida mt-2 for catabolism of m-xylene is an archetypal model for environmental biodegradation of aromatic pollutants.
Although nearly every metabolic and transcriptional component of this regulatory system
is known to an extraordinary molecular detail, the complexity of its architecture
is still perplexing.
The genetic network of the TOL plasmid pWW0 of the soil bacterium Pseudomonas putida mt-2 for catabolism of m-xylene is an archetypal model for environmental biodegradation of aromatic pollutants.
Although nearly every metabolic and transcriptional component of this regulatory system
is known to an extraordinary molecular detail, the complexity of its architecture
is still perplexing.
To gain an insight into the inner layout of this network a logic
model of the TOL system was implemented, simulated and experimentally validated. This
analysis made sense of the specific regulatory topology out on the basis of an unprecedented
network motif around which the entire genetic circuit for m-xylene catabolism gravitates.


